Vorbereitung
Laden Sie zunächst den Datensatz
fb25von der pandaR-Website durch die bekannten Befehle direkt ins Environment. Alternativ ist die Datei unter diesem Link zum Download verfügbar. Beachten Sie in jedem Fall, dass die Ergänzungen im Datensatz vorausgesetzt werden. Die Bedeutung der einzelnen Variablen und ihre Antwortkategorien können Sie dem Dokument Variablenübersicht entnehmen.
R-Code für die Vorbereitung
#### Was bisher geschah: ----
# Daten laden
load(url('https://pandar.netlify.app/daten/fb25.rda'))
# Nominalskalierte Variablen in Faktoren verwandeln
fb25$hand_factor <- factor(fb25$hand,
levels = 1:2,
labels = c("links", "rechts"))
fb25$fach <- factor(fb25$fach,
levels = 1:5,
labels = c('Allgemeine', 'Biologische', 'Entwicklung', 'Klinische', 'Diag./Meth.'))
fb25$ziel <- factor(fb25$ziel,
levels = 1:4,
labels = c("Wirtschaft", "Therapie", "Forschung", "Andere"))
fb25$wohnen <- factor(fb25$wohnen,
levels = 1:4,
labels = c("WG", "bei Eltern", "alleine", "sonstiges"))
Falls Sie nochmal sicher gehen wollen, ob alles korrekt funktioniert hat, könnte die Anzahl der Zeilen und Spalten einen Hinweis geben:
dim(fb25)
## [1] 211 44
Der Datensatz besteht aus 211 Zeilen (Beobachtungen) und 44 Spalten (Variablen). Falls Sie bereits eigene Variablen erstellt haben, kann die Spaltenzahl natürlich abweichen.
Aufgabe 1
Erstellen Sie im Datensatz fb25 die Skalenwerte für die Subskala ruhig/unruhig der aktuellen Stimmung, die mit den Items mdbf3, mdbf6, mdbf9 und mdbf12 gemessen wurde. mdbf3 und mdbf9 sind invertiert und müssen rekodiert werden. Speichern sie diese als ru_pre im Datensatz fb25 ab.
- Erstellen Sie den Skalenwert als Mittelwert der vier Items.
Lösung
# Invertieren
fb25$mdbf3_r <- -1 * (fb25$mdbf3 - 5)
fb25$mdbf9_r <- -1 * (fb25$mdbf9 - 5)
# Skalenwert
ru_pre <- fb25[, c("mdbf3_r", "mdbf6", "mdbf9_r", "mdbf12")]
fb25$ru_pre <- rowMeans(ru_pre)
Oder in einem Schritt mit der Pipe:
# Skalenwert
fb25$ru_pre <- fb25[, c("mdbf3_r", "mdbf6",
"mdbf9_r", "mdbf12")] |> rowMeans()
Aufgabe 2
Bestimmen Sie für die Prokrastination, fb25$prok den gesamten Mittelwert und Median.
- Was vermuten Sie, aufgrund des Verhältnisses der beiden Maße der zentralen Tendenz, bezüglich der Schiefe der Verteilung?
- Prüfen Sie Ihre Vermutung anhand eines Histogramms!
Lösung
# Median und Mittelwert
median(fb25$prok, na.rm = TRUE)
## [1] 2.7
mean(fb25$prok, na.rm = TRUE)
## [1] 2.7
Der Median ist gleich dem Mittelwert, was eine symmetrische Verteilung vermuten lässt.
Prüfen der Vermutung anhand eines Histogramms!
hist(fb25$prok, breaks = 8) # Histogramm
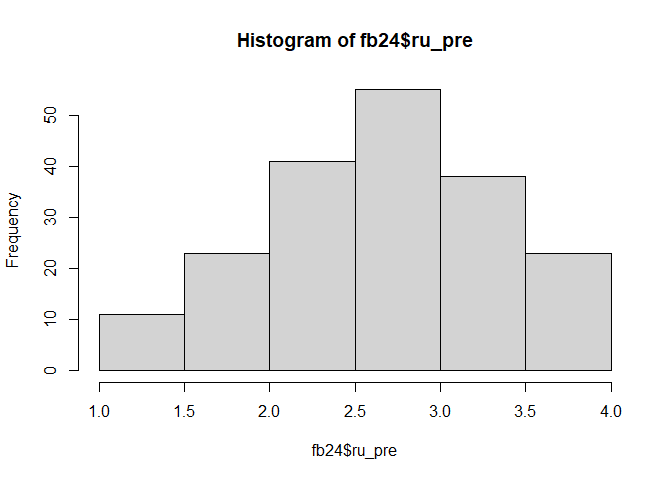
Unser Histogramm zeigt uns, dass die Verteilung tatsächlich einigermaßen symmetrisch ist.
Aufgabe 3
Bestimmen Sie für den Skalenwert ru_pre die empirische Varianz und Standardabweichung. Achten Sie dabei darauf, ob es auf der Skala fehlende Werte gibt.
- Sind empirische Varianz und Standardabweichung größer oder kleiner als diejenige Schätzung, die mithilfe von
var()odersd()bestimmt wird?
Lösung
Erinnerung:
- Empirische Varianz: $s^2_{X} = \frac{\sum_{m=1}^n (x_m - \bar{x})^2}{n}$
- Schätzer der Populationsvarianz: $\hat{\sigma}^2_{X} = \frac{\sum_{m=1}^n (x_m - \bar{x})^2}{n - 1}$
Zur Berechnung der Varianz gemäß Formel benötigen wir $n$. Wir könnten mit nrow(fb25) die Länge des Datensatzes für n heranziehen. Dies ist jedoch nur dann sinnvoll, wenn auf der Variable `ru_pre`` keine fehlenden Werte vorhanden sind!
is.na(fb25$ru_pre) |> sum()
## [1] 0
Es gibt keine fehlenden Werte, daher benötigt man weder na.omit um die NAs auszschließen, noch na.rm=T als Argument für den Mittelwert. Es bringt, außer zusätzlicher Komputationen, jedoch auch keinen Nachteil diese Argumente stehen zu lassen. Nachfolgend sind sie mit aufgeführt um den flexibelsten Fall zu demonstrieren:
# empirische Varianz
# per Hand
sum((fb25$ru_pre - mean(fb25$ru_pre, na.rm = T))^2, na.rm = T) / (length(na.omit(fb25$ru_pre)))
## [1] 0.4449445
# durch Umrechnung
var(fb25$ru_pre, na.rm = T) * (length(na.omit(fb25$ru_pre))-1) / length(na.omit(fb25$ru_pre))
## [1] 0.4449445
# Populationsschätzer
var(fb25$ru_pre, na.rm = T)
## [1] 0.4470633
Die empirische Varianz ist kleiner als der Populationsschätzer.
Nun fehlt noch die Betrachtung der Standardabweichung. Als einfachste Möglichkeit für die Berechnung der empirischen Standardabweichung haben wir gelernt, dass man die Wurzel aus der empirischen Varianz ziehen kann.
# empirische Standardabweichung (na.omit / na.rm kann auch ausgelassen werden!)
(sum((fb25$ru_pre - mean(fb25$ru_pre, na.rm = T))^2, na.rm = T) / length(na.omit(fb25$ru_pre))) |> sqrt()
## [1] 0.6670416
# Populationsschätzer
sd(fb25$ru_pre, na.rm = T)
## [1] 0.6686279
Auch hier ist der empirische Wert kleiner als der Schätzer.
Aufgabe 4
Erstellen Sie eine z-standardisierte Variante der Skala ruhig/unruhig und legen Sie diese im Datensatz als neue Variable ru_pre_zstd an.
- Erstellen Sie für die z-standardisierte Variable ein Histogramm.
- Was fällt Ihnen auf, wenn Sie dieses mit dem Histogramm der unstandardisierten Werte (Erinnerung: Variablennamen
ru_pre) vergleichen? - Erstellen Sie beide Histogramme noch einmal mit 40 angeforderten Breaks.
Lösung
Um die Vergleichbarkeit zu erhöhen, wird im folgenden Code ein kleiner Trick angewendet. Die beiden Histogramme sollten am besten gleichzeitig unter Plots angezeigt werden. Durch die verwendete Funktion par() kann man verschiedene Plots gemeinsam in einem Fenster zeichnen. Das Argument bestimmt dabei, dass es eine Zeile und zwei Spalten für die Plots gibt.
par(mfrow=c(1,2))
# z-Standardisierung
fb25$ru_pre_zstd <- scale(fb25$ru_pre, center = TRUE, scale = TRUE)
# Histogramme
hist(fb25$ru_pre_zstd)
hist(fb25$ru_pre)
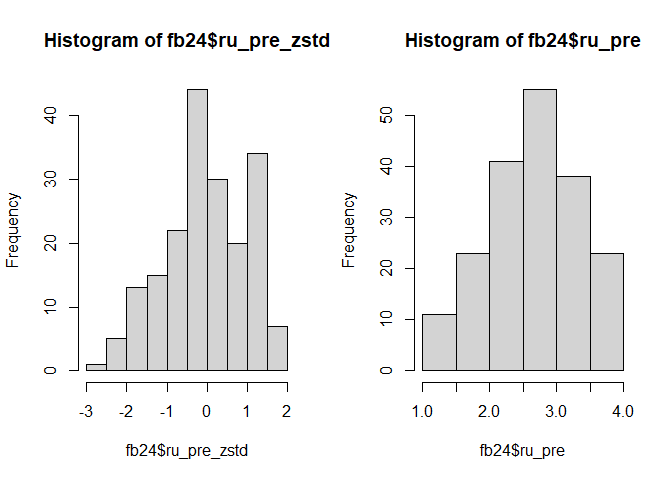
Beim Vergleich der beiden Histogrammen fällt auf, dass sich - aufgrund der R-Voreinstellungen - das Erscheinungsbild fälschlicherweise unterscheidet (vor allem, wenn wir die y-Achse betrachten!) - eigentlich sollte sich durch die z-Transformation nur Skalierung der x-Achsen-Variable verändern. Tatsächlich aber bestimmt R hier eine unterschiedliche Anzahl von Kategorien. Wir erhalten eine konstantere Darstellung durch das breaks-Argument:
# Histogramme mit jeweils 5/6 Breaks
par(mfrow=c(1,2))
hist(fb25$ru_pre_zstd, breaks = 5)
hist(fb25$ru_pre, breaks = 6)
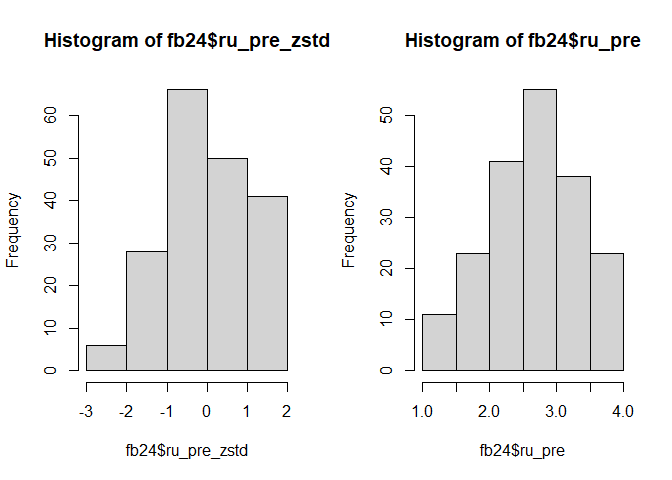
Die Verteilungen sehen nun vergleichbarer aus. Da die Breaks ein weicher Befehl sind, kann hier keine komplette Gleichheit gegeben werden.
Zum Abschluss sollte noch der Grafik-Bereich wieder so eingestellt werden, dass nur eine Grafik gleichzeitig angezeigt wird.
par(mfrow=c(1,1))
](/media/header/frogs_on_phones.jpg)