Kernfragen dieser Lehreinheit über Gruppenvergleiche
- Wie fertige ich Deskriptivstatistiken (Grafiken, Kennwerte) zur Veranschaulichung des Unterschieds zwischen zwei Gruppen an?
- Was sind Voraussetzungen des t-Tests und wie prüfe ich sie?
- Wie führe ich einen t-Test in R durch?
- Wie berechne ich die Effektstärke Cohen’s d?
- Wie führe ich den Wilcoxon-Test (auch “Mann-Whitney-Test”, “U-Test”, “Mann-Whitney-U-Test”, “Wilcoxon-Rangsummentest”) in R durch?
Vorbereitende Schritte
Den Datensatz fb23 haben wir bereits über diesen Link heruntergeladen und können ihn über den lokalen Speicherort einladen oder Sie können Ihn direkt mittels des folgenden Befehls aus dem Internet in das Environment bekommen. In den vorherigen Tutorials und den dazugehörigen Aufgaben haben wir bereits Änderungen am Datensatz durchgeführt, die hier nochmal aufgeführt sind, um den Datensatz auf dem aktuellen Stand zu haben:
#### Was bisher geschah: ----
# Daten laden
load(url('https://pandar.netlify.app/daten/fb23.rda'))
# Nominalskalierte Variablen in Faktoren verwandeln
fb23$hand_factor <- factor(fb23$hand,
levels = 1:2,
labels = c("links", "rechts"))
fb23$fach <- factor(fb23$fach,
levels = 1:5,
labels = c('Allgemeine', 'Biologische', 'Entwicklung', 'Klinische', 'Diag./Meth.'))
fb23$ziel <- factor(fb23$ziel,
levels = 1:4,
labels = c("Wirtschaft", "Therapie", "Forschung", "Andere"))
fb23$wohnen <- factor(fb23$wohnen,
levels = 1:4,
labels = c("WG", "bei Eltern", "alleine", "sonstiges"))
# Rekodierung invertierter Items
fb23$mdbf4_pre_r <- -1 * (fb23$mdbf4_pre - 4 - 1)
fb23$mdbf11_pre_r <- -1 * (fb23$mdbf11_pre - 4 - 1)
fb23$mdbf3_pre_r <- -1 * (fb23$mdbf3_pre - 4 - 1)
fb23$mdbf9_pre_r <- -1 * (fb23$mdbf9_pre - 4 - 1)
# Berechnung von Skalenwerten
fb23$gs_pre <- fb23[, c('mdbf1_pre', 'mdbf4_pre_r',
'mdbf8_pre', 'mdbf11_pre_r')] |> rowMeans()
fb23$ru_pre <- fb23[, c("mdbf3_pre_r", "mdbf6_pre",
"mdbf9_pre_r", "mdbf12_pre")] |> rowMeans()
# z-Standardisierung
fb23$ru_pre_zstd <- scale(fb23$ru_pre, center = TRUE, scale = TRUE)
Was erwartet Sie?
Nachdem wir uns zuletzt mit dem Unterschied zwischen dem Mittelwert einer Stichprobe und einem von uns theoretisch postulierten Wert ($\mu_0$) auseinandergesetzt haben, fokussieren wir uns nun auf Unterschiede zwischen zwei Gruppen. Hierbei muss zwischen unabhängigen und abhängigen Stichproben unterschieden werden - um den ersten Fall geht es uns hier, den Zweiten gucken wir uns in der nächsten Sitzung an.
In dieser Sitzung geht es uns um primär um zwei Vergleiche:
- Erreichen klinisch und nicht klinisch interessierte Studierende in der Verträglichkeit im Durchschnitt gleich hohe Werte? (t-Test und Cohen’s d für unabhängige Stichproben)
- Erreichen klinisch interessierte im Gegensatz zu nicht klinisch interessierten Studierenden in der Lebenszufriedenheit im Mittel höhere Werte? (Wilcoxon-Test für unabhängige Stichproben)
Außerdem finden Sie im Anhang auch noch eine weitere Frage:
- Haben Studierende mit Wohnort in Uninähe (Frankfurt) mit gleicher Wahrscheinlichkeit einen Nebenjob wie Studierende, deren Wohnort außerhalb von Frankfurt liegt? (Vierfelder-$\chi^2$-Test)
Mittelwertsvergleich
Wie oben bereits vorgestellt, geht es uns in diesem Abschnitt darum, dass wir untersuchen wollen, ob Studierende, die sich für die klinische Psychologie interessieren, im Mittel das gleiche Ausmaß an Verträglichkeit aufweisen wie Studierende, deren Hauptinteresse in einem der vielen und spannenden anderen Bereiche der Psychologie liegt. Hinsichtlich der Richtung der Hypothese können wir uns nicht genau festlegen, weil wir für beides hinreichend gute Argumente gefunden haben. Haben Personen, die sich für die klinische Psychologie interessieren eine natürlich Faszination für Störungen des Verhaltens und sind deswegen weniger verträglich als andere Personen ($H_1: \mu_1 < \mu_2$)? Oder tendieren Studierende mit einem Interesse für die klinische Psychologe dazu, anderen bei der Bewältigung ihrer Hürden helfen zu wollen und sind daher verträglicher ($H_1: \mu_1 > \mu_2$)? Weil beides gleichermaßen plausibel erscheint, legen wir ein ungerichtetes Hypothesenpaar fest: $H_0 : \mu_1 = \mu_2$ und $H_1: \mu_1 \neq \mu_2$.
Vorbereitende Schritte
Die Gruppierungsvariable in unserer Fragestellung muss für die Nutzung der Funktionen in R als Faktor vorliegen. Bei uns soll es um das Interesse am Fach klinischer Psychologie gehen (ja vs. nein). Die entsprechende (nominalskalierte) Variable liegt noch nicht im Datensatz vor, kann aber einfach erstellt werden, indem die nichtklinischen Interessen in eine Kategorie zusammengefasst werden. Dies erfolgt per logischer Abfrage (fb23$fach == "Klinische") und anschließender Transformation des Ergebnisses in eine Faktorvariable:
fb23$fach_klin <- factor(as.numeric(fb23$fach == "Klinische"),
levels = 0:1,
labels = c("nicht klinisch", "klinisch"))
Wir können mit is.factor() überprüfen, ob die Variable fach_klin im Datensatz tatsächlich ein Faktor ist.
is.factor(fb23$fach_klin)
## [1] TRUE
Diese Funktion gibt uns einen boolschen Wert (TRUE oder FALSE) und beantwortet damit unsere Frage, ob das klinische Interesse ein factor ist oder nicht. Wir sehen aufgrund der Antwort als TRUE, dass die Variable fach_klin als Faktor vorliegt.
Auch die levels können wir bestimmen
levels(fb23$fach_klin)
## [1] "nicht klinisch" "klinisch"
Der Faktor hat also 2 Stufen.
Deskriptivstatistik
Im ersten Schritt wollen wir uns die Daten deskriptiv anschauen. Dazu können wir die Daten entweder visuell oder durch statistische Kennwerte aufbereiten. Wir werfen zunächst vorbereitend einen tabellarischen Blick auf die Variable klinisches Interesse:
table(fb23$fach_klin)
##
## nicht klinisch klinisch
## 85 82
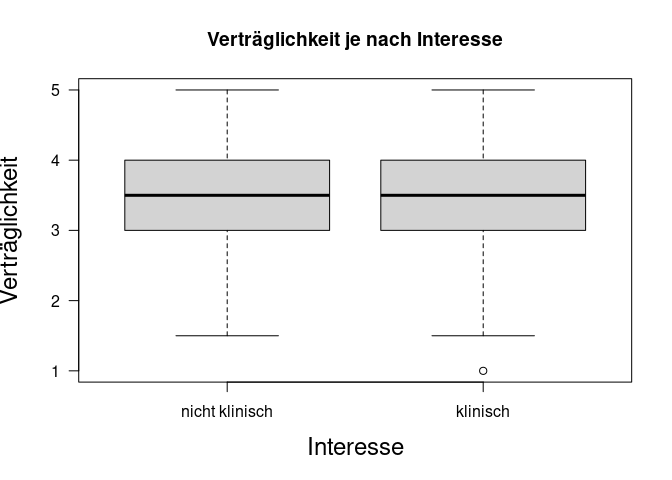
Mithilfe eines Boxplots, bzw. mithilfe von Histogrammen, lassen sich Unterschiede zwischen Gruppen gut untersuchen. Wir verwenden hier einige Zusatzeinstellungen, um die Grafiken übersichtlicher und auch ein wenig ansprechender zu gestalten.
# Gruppierter Boxplot :
boxplot(fb23$vertr ~ fb23$fach_klin,
xlab="Interesse", ylab="Verträglichkeit",
las=1, cex.lab=1.5,
main="Verträglichkeit je nach Interesse")
# Je ein Histogramm pro Gruppe, untereinander dargestellt, vertikale Linie für den jeweiligen Mittelwert
par(mfrow=c(2,1), mar=c(3,2,2,0))
hist(fb23[(fb23$fach_klin=="nicht klinisch"), "vertr"], main="(nicht klinisch)",
xlim=c(0,6), xlab="", ylab="", las=1)
abline(v=mean(fb23[(fb23$fach_klin=="nicht klinisch"), "vertr"], na.rm=T), col="aquamarine3", lwd=3)
hist(fb23[(fb23$fach_klin=="klinisch"), "vertr"], main="(klinisch)",
xlim=c(0,6), xlab="", ylab="", las=1)
abline(v=mean(fb23[(fb23$fach_klin=="klinisch"), "vertr"], na.rm=T), col="darksalmon", lwd=3)
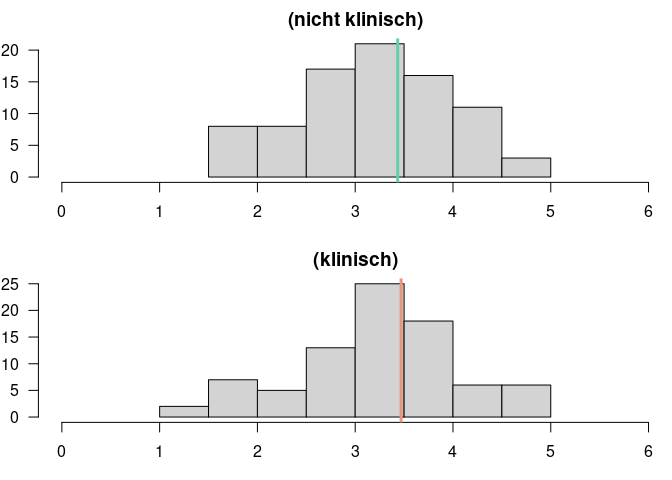
fb23$vertr ~ fb23$fach_klin ist die Formelnotation in R, welche Sie im Rahmen der Regression noch genauer kennenlernen werden. Links von der ~ (Tilde) steht die abhängige Variable (hier der Wert in der Verträglichkeit), deren Mittelwertsunterschiede durch die unabhängige Variable (hier Interesse) rechts der ~ “erklärt” werden soll. Der Befehl par(mfrow=c(2,1), mar=c(3,2,2,0)) bewirkt, dass 2 Grafiken untereinander dargestellt werden - und zwar im selben Plot. Das Argument mfrow definiert ein Plot-Array mit der angegebenen Anzahl von Zeilen und Spalten (wobei mit mfrow die Plots zeilenweise verteilt werden; alternativ mit mfcol). Mit dem Argument mar werden für unten, links, oben und rechts die Randabstände spezifiziert. Spielen Sie doch einmal selbst an den Einstellungen herum und schauen nach, was die Argumente jeweils bewirken!
Damit von nun an nicht immer zwei Grafiken in einem Plot dargestellt werden, können wir die Einstellungen folgendermaßen zurücksetzen:
dev.off()
## null device
## 1
Wir können uns auch Deskriptivstatistiken ansehen. Bspw. könnten wir uns die Mittelwerte oder die SDs etc. ausgeben lassen. Dazu nehmen wir entweder die summary() und wählen die entsprechenden Fälle aus oder wir machen uns das psych-Paket zunutze. Wir hatten im letzen Beitrag detaillierter besprochen, was Pakete sind und wie sie funktionieren. Um psych nutzen zu können, muss es installiert sein (install.packages()). Falls dem so ist, kann das Paket mit library() eingeladen werden. Die Funktion, die uns interessiert, heißt describeBy(), welche die Gruppenaufteilung bereits für uns übernimmt.
library(psych)
##
## Attaching package: 'psych'
## The following objects are masked from 'package:ggplot2':
##
## %+%, alpha
describeBy(x = fb23$vertr, group = fb23$fach_klin) # beide Gruppen im Vergleich
##
## Descriptive statistics by group
## group: nicht klinisch
## vars n mean sd median trimmed mad min max range skew kurtosis se
## X1 1 84 3.43 0.8 3.5 3.46 0.74 1.5 5 3.5 -0.19 -0.58 0.09
## --------------------------------------------------------------------------------------
## group: klinisch
## vars n mean sd median trimmed mad min max range skew kurtosis se
## X1 1 82 3.47 0.85 3.5 3.5 0.74 1 5 4 -0.4 0.1 0.09
Achtung, bei den hier berichteten sd handelt es sich nicht um die Stichprobenkennwerte, sondern um die Populationsschätzer. Daher berechnen wir die Standardabweichung auch nochmals per Hand:
vertr_nichtKlinisch <- fb23$vertr[fb23$fach_klin=="nicht klinisch"]
sigma_nichtKlinisch <- sd(vertr_nichtKlinisch, na.rm = T)
n_nichtKlinisch <- length(vertr_nichtKlinisch[!is.na(vertr_nichtKlinisch)])
sd_nichtKlinisch <- sigma_nichtKlinisch * sqrt((n_nichtKlinisch-1) / n_nichtKlinisch)
sd_nichtKlinisch
## [1] 0.7972414
vertr_klinisch <- fb23$vertr[fb23$fach_klin=="klinisch"]
sigma_klinisch <- sd(vertr_klinisch, na.rm = T)
n_klinisch <- length(vertr_klinisch[!is.na(vertr_klinisch)])
sd_klinisch <- sigma_klinisch * sqrt((n_klinisch-1) / n_klinisch)
sd_klinisch
## [1] 0.8422804
Voraussetzungen
Damit wir den Ergebnissen des t-Tests trauen können, müssen dessen Voraussetzungen erfüllt sein. Diese sind:
- zwei unabhängige Stichproben $\rightarrow$ ok
- die einzelnen Messwerte innerhalb der Gruppen sind voneinander unabhängig (Messwert einer Vpn hat keinen Einfluss auf den Messwert einer anderen) $\rightarrow$ ok
- das untersuchte Merkmal ist in den Grundgesamtheiten der beiden Gruppen normalverteilt $\rightarrow$ (ggf.) optische Prüfung
- Homoskedastizität: Varianzen der Variablen innerhalb der beiden Populationen sind gleich $\rightarrow$ Levene-Test
Wenn das Merkmal, das wir untersuchen, in der Population normalverteilt ist (Voraussetzung 3), können wir den $t$-Test nutzen. Allerdings haben wir in den meisten Fällen keine zusätzliche Information über die Verteilung in der Population als die, die wir aus unserer Stichprobe ziehen können. Daher ist es üblich, die Annahme optisch zu prüfen. Das werden wir im Folgenden auch tun, nur vorab der Zusatz, dass in vielen Studien aufgrund der Stichprobengröße auf die Prüfung der Normalverteilungsannahme verzichtet wird. In Fällen, in denen die Stichprobe ausreichend groß ist, folgt die Stichprobenkennwerteverteilung wegen des zentralen Grenzwertsatzes auch unabhängig von der Verteilung in der Population der $t$-Verteilung. Dabei ist es Auslegungssache was genau “ausreichend groß” bedeutet. Häufig wird die Daumenregel von $n > 30$ in jeder Gruppe genutzt. Bei solch kleinen Stichproben greift der Effekt allerdings nur dann, wenn das Merkmal zumindest symmetrisch verteilt ist. Andere Empfehlungen gehen besonders bei sehr schiefen Verteilungen in Richtung von $n > 80$ pro Gruppe.
Um die Verteilung optisch zu prüfen, haben wir zwei Möglichkeiten:
- Möglichkeit 1: die bei Normalverteilung erwartete Dichtefunktion über das Histogramm legen und so die Übereinstimmung beurteilen
- Möglichkeit 2: QQ-Plot (quantile-quantile): es wird die beobachtete Position eines Messwerts gegen diejenige Position, die unter Gültigkeit der Normalverteilung zu erwarten wäre, abgetragen. Bei Normalverteilung liegen die Punkte (in etwa) auf einer Geraden.
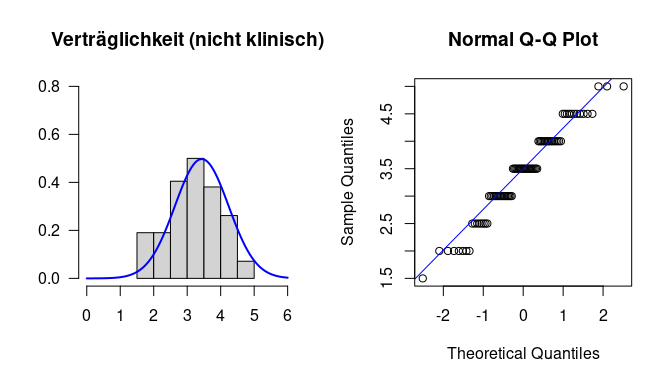
# Gruppe 1 (nichtKlinisch)
par(mfrow=c(1,2))
vertr_nichtKlinisch <- fb23[(fb23$fach_klin=="nicht klinisch"), "vertr"]
hist(vertr_nichtKlinisch,
xlim=c(0,6), ylim=c(0,.8),
main="Verträglichkeit (nicht klinisch)",
xlab="", ylab="",
las=1, probability=T)
curve(dnorm(x,
mean = mean(vertr_nichtKlinisch, na.rm=T),
sd = sd(vertr_nichtKlinisch, na.rm=T)),
col="blue", lwd=2, add=T)
qqnorm(vertr_nichtKlinisch)
qqline(vertr_nichtKlinisch, col="blue")
dev.off()
## null device
## 1
Mithilfe von curve() kann eine Linie in eine Grafik eingezeichnet werden. Hierbei bezeichnet x die x-Koordinate. dnorm() hatten wir bereits kennen gelernt. Diese Funktion beschreibt die Dichte der Normalverteilung. Die Normalverteilung ist eindeutig durch ihren Mittelwert und durch ihre Standardabweichung definiert. Wir müssen dnorm() also jeweils den empirischen Mittelwert sowie die empirische Standardabweichung übergeben. Wenn man es sehr genau nehmen will, müsste man hier also eine Verrechnung der von R erzeugten Standardabweichung vornehmen. Wie im letzten Tutorial beschrieben nutzen wir aber einfach das Ergebnis von sd(). Dies wird auch in allen weiteren Tutorials ohne zusätzlichen Hinweis gemacht. Das Argument add = T ist nötig, da sonst ein neuer Plot für die Kurve erstellt wird. Durch add = T wird sie dem Histogramm hinzugefügt. Damit die Dichte sichtbar ist, muss im Histogramm zuvor das Argument probability = T gewählt werden. Ansonsten würden absolute Häufigkeiten anstatt der relativen Häufigkeiten abgetragen werden. Den qqnorm()-Befehl hatten wir auch bereits kennen gelernt. Mit qqline() erhalten wir die nötige Linie, auf welcher die Punkte einigermaßen liegen müssen, damit sie als normalverteilt einzustufen sind.
Die Treppenform, die wir hier im QQ-Plot sehen ist für fragebogenbasierte Daten sehr typisch. Sie entsteht dadurch, dass wir mit vorgegebenen Antwortkategorien keine stetige Variable erzeugen können. Stattdessen gibt es hier anscheinend nur acht verschiedene Ausprägungen der Variable vertr. Dadurch, dass dieser Skalenwert als Mittelwert aus zwei fünfstufigen Items erzeugt wurde sind theoretisch nur neun Ausprägungen möglich - alle Zahlen von 1 bis 5 in 0,5er Schritten. Je mehr Items wir zusammenführen, um einen Skalenwert zu berechnen, desto besser können wir eine stetige Variable approximieren. Für die Prokrastinationstendenz prok z.B. haben wir einen Skalenwert aus zehn verschiedenen Items mit jeweils vier Antwortkategorien erzeugt, sodass wir 31 mögliche Ausprägungen haben.
Abgesehen von dieser Treppenform scheinen die Punkte hier aber relativ nahe an der Linie zu liegen, sodass wir zunächst zumindest nicht von starker Verletzung der Normalverteilungsannahme ausgehen müssen. In Kombination mit der Tatsache, dass wir in der Gruppe 84 Personen beobachtet haben, können wir hier den $t$-Test nutzen.
Wir wiederholen die Befehle von zuvor auch für die zweite Gruppe:
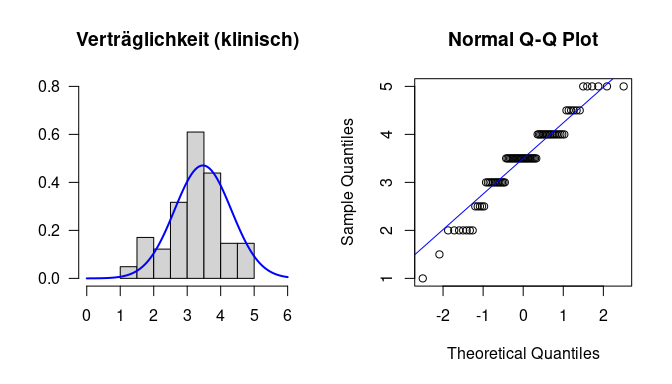
# Gruppe 2 (klinisch)
par(mfrow=c(1,2))
vertr_klinisch <- fb23[(fb23$fach_klin=="klinisch"), "vertr"]
hist(vertr_klinisch,
xlim=c(0,6), ylim=c(0,.8),
main="Verträglichkeit (klinisch)",
xlab="", ylab="", las=1, probability=T)
curve(dnorm(x,
mean = mean(vertr_klinisch, na.rm=T),
sd = sd(vertr_klinisch, na.rm=T)),
col="blue", lwd=2, add=T)
qqnorm(vertr_klinisch)
qqline(vertr_klinisch, col="blue")
dev.off()
## null device
## 1
In dieser Gruppe sehen wir eine etwas stärkere Schiefe, als in der ersten Gruppe. Das wird besonders im unteren Wertebereich deutlich, wo wir mehr Personen beobachten, als wir unter der Normalverteilung erwarten würden (wie im Histogramm zu sehen ist). In die Logik des QQ-Plots übersetzt heißt das, dass die untersten Personen in der Rangreihe von Werte niedrigere Werte haben, als wir unter der Normalverteilung erwarten würden - die Punkte links liegen unter der Gerade. Dennoch können wir auch hier - unter Berücksichtigung der Tatsache, dass wir 82 Personen in dieser Gruppe haben - davon ausgehen, dass die Teststatistik ausreichend gut der $t$-Verteilung folgen sollte.
Die vierte Annahme (Homoskedastizität - also die Varianzgleichheit in beiden Populationen) wird typischerweise mit einem inferenzstatistischen Verfahren geprüft. Der Test heißt Levene-Test und das Hypothesenpaar, das er prüft sieht so aus:
- $H_0$: $\sigma_1 = \sigma_2$ (Homoskedastizität ist gegeben)
- $H_1$: $\sigma_1 \neq \sigma_2$ (Homoskedastizität ist nicht gegeben)
Ein nicht-signifikantes Ergebnis (p > .05) deutet also darauf hin, dass wir nicht ausreichend Grundlage haben, um die Homoskedastizitätsannahme zu verwerfen. Den Levene-Test können wir mit Hilfe des car-Pakets durchführen (dieses muss natürlich vorher installiert sein). Die nötige Funktion heißt leveneTest(). Sie nimmt eine Formel entgegen, die die Punktzahl ihrer jeweiligen Gruppe zuweist. Links von der ~ (Tilde) steht die abhängige Variable (hier der Verträglichkeitswert), deren Mittelwertsunterschied durch die unabhängige Variable (hier Interesse) rechts der ~ erklärt werden soll. Wir merken uns also, dass die Gruppenzugehörigkeit rechts der ~ stehen muss.
library(car)
## Loading required package: carData
##
## Attaching package: 'car'
## The following object is masked from 'package:psych':
##
## logit
leveneTest(fb23$vertr ~ fb23$fach_klin)
## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 1 0.0118 0.9135
## 164
Das Testergebnis zeigt mit einem $p$-Wert von 0.913, dass die Nullhypothese beibehalten wird, wir also weiterhin von der Gültigkeit der Homoskedastizitätsannahme ausgehen können. Für den Fall, dass die Annahme nicht hält, gibt es die Welch-Korrektur für den $t$-Test. Diese ist in R sogar die Voreinstellung.
Durchführung des $t$-Test
Zur Erinnerung - unsere Fragestellung für diesen Test war:
Erreichen klinisch und nicht klinisch interessierte Studierende in der Verträglichkeit im Durchschnitt gleich hohe Werte?
Weil die Fragestellung ungerichtet ist, brauchen wir auch eine ungerichtete Hypothese. Konkret ist unsere Hypothese also eine ungerichtete Unterschiedshypothese. Inhaltlich lässt sich das Paar aus $H_0$ und $H_1$ so formulieren:
- $H_0$: Klinisch und nicht klinisch interessierte Studierende unterscheiden sich nicht im Verträglichkeitswert.
- $H_1$: Klinisch und nicht klinisch interessierte Studierende unterscheiden sich im Verträglichkeitswert.
Etwas formaler ausgedrückt:
- $H_0$: $\mu_\text{nicht klinisch} = \mu_\text{klinisch}$ bzw. $\mu_\text{nicht klinisch} - \mu_\text{klinisch} = 0$
- $H_1$: $\mu_\text{nicht klinisch} \ne \mu_\text{klinisch}$ bzw. $\mu_\text{nicht klinisch} - \mu_\text{klinisch} \ne 0$
Wie so häufig gehen wir von einem $\alpha$-Fehlerniveau von 5% aus.
Wir hatten im Rahmen des Einstichproben-t-Tests bereits die Funktion t.test() kennengelernt. Diese nutzen wir wieder. Wir übergeben dieser wieder die Formel, die wir bereits im Boxplot und im Levene-Test verwendet haben. Außerdem wählen wir einige Zusatzargumente, die dann zum Zweistichproben-t-Test für unabhängige Stichproben führen:
t.test(fb23$vertr ~ fb23$fach_klin, # abhängige Variable ~ unabhängige Variable
paired = FALSE, # Stichproben sind unabhängig
alternative = "two.sided", # zweiseitige Testung (Default)
var.equal = TRUE, # Homoskedastizität liegt vor (-> Levene-Test)
conf.level = .95) # alpha = .05 (Default)
##
## Two Sample t-test
##
## data: fb23$vertr by fb23$fach_klin
## t = -0.27326, df = 164, p-value = 0.785
## alternative hypothesis: true difference in means between group nicht klinisch and group klinisch is not equal to 0
## 95 percent confidence interval:
## -0.2878085 0.2178318
## sample estimates:
## mean in group nicht klinisch mean in group klinisch
## 3.434524 3.469512
Anhand der Ergebnisse können wir folgende Aussage treffen: Studierende, die sich für klinische Inhalte interessieren unterscheiden sich in ihrer Verträglichkeit nicht bedeutsam von Studierenden, die sich primäre für andere Inhalte interessieren ($t = 164, df = -0.273$, $p = 0.785$).
Berechnung der Effektstärke Cohen’s $d$
Wir wissen nun, dass es zwischen Populationen, aus denen die beiden Stichproben kommen keinen Unterschied in den Erwartungswerten gibt. Dennoch wollen wir zur Eindordnung des Ergebnisses die Effektstärke ermitteln: Cohen’s $d$ gibt den standardisierten Mittelwertsunterschied zwischen zwei Gruppen an. “Standardisiert” bedeutet, dass wir uns nicht mehr auf der Originalmetrik befinden (hier auf der Skala von 1 bis 5), sondern mit Standardabweichungen arbeiten. Ein Wert von 1 zeigt also an, dass sich die Gruppenmittelwerte um eine Standardabweichung voneinander unterscheiden. Die Effektstärke berechnet sich wie folgt:
\begin{equation} \small d = \frac{\bar{x}_1-\bar{x}_2}{\hat{\sigma}_{inn}} \end{equation}wobei
\begin{equation} \small \hat{\sigma}_{inn} = \sqrt{\frac{{\hat{\sigma}_1^2}\cdot(n_1-1) + {\hat{\sigma}^2_2}\cdot(n_2-1)} {(n_1-1) + (n_2-1)}} \end{equation}Cohen (1988) hat folgende Konventionen zur Beurteilung der Effektstärke $d$ vorgeschlagen, die man heranziehen kann, um den Effekt “bei kompletter Ahnungslosigkeit” einschätzen zu können (wissen wir mehr über den Sachverhalt, so sollten Effektstärken lieber im Bezug zu anderen Studienergebnissen interpretiert werden):
| $d$ | Interpretation |
|---|---|
| ~ .2 | kleiner Effekt |
| ~ .5 | mittlerer Effekt |
| ~ .8 | großer Effekt |
Wir führen die Berechnung von Cohen’s $d$ zunächst von Hand durch. Dafür speichern wir uns die nötigen Größen ab und wenden dann die präsentierte Formel an:
vertr_nichtKlinisch <- fb23[(fb23$fach_klin=="nicht klinisch"), "vertr"]
mw_nichtKlinisch <- mean(vertr_nichtKlinisch, na.rm=T)
n_nichtKlinisch <- length(vertr_nichtKlinisch[!is.na(vertr_nichtKlinisch)])
sigma_qu_nichtKlinisch <- var(vertr_nichtKlinisch, na.rm=T)
vertr_klinisch <- fb23[(fb23$fach_klin=="klinisch"), "vertr"]
mw_klinisch <- mean(vertr_klinisch, na.rm=T)
n_klinisch <- length(vertr_klinisch[!is.na(vertr_klinisch)])
sigma_qu_klinisch <- var(vertr_klinisch, na.rm=T)
sigma_inn <- sqrt((sigma_qu_nichtKlinisch* (n_nichtKlinisch - 1) + sigma_qu_klinisch* (n_klinisch - 1)) / (n_nichtKlinisch-1 + n_klinisch-1))
d1 <- (mw_nichtKlinisch - mw_klinisch) / sigma_inn
d1
## [1] -0.04242135
Natürlich gibt es in R auch eine angenehmere Alternative:
# install.packages("effsize")
library("effsize")
##
## Attaching package: 'effsize'
## The following object is masked from 'package:psych':
##
## cohen.d
d2 <- cohen.d(fb23$vertr, fb23$fach_klin, na.rm=T)
d2
##
## Cohen's d
##
## d estimate: -0.04242135 (negligible)
## 95 percent confidence interval:
## lower upper
## -0.3489853 0.2641426
Der Vorteil der Funktion cohen.d ist, dass wir zusätzlich zur Effektstärke direkt auch ein Konfidenzintervall erhalten. Der Wert sollte aber natürlich der Gleiche sein, wie der, den wir gerade per Hand berechnet hatten.
Ergebnisinterpretation
Wir haben untersucht, ob sich klinisch und nicht klinisch interessierte Studierende im Wert Ihrer Verträglichkeit unterscheiden. Tatsächlich findet sich deskriptiv ein nur geringer Unterschied: Nicht klinisch interessierte Studierende weisen einen durchschnittlichen Wert von 3.435 (SD = 0.797) auf, während klinisch interessierte Studierende einen Wert von 3.47 (SD = 0.842) aufweisen. Dies entspricht nach Cohens Konvention (1988) einem sehr kleinen Effekt ($d $= -0.042).
Zur Beantwortung der Fragestellung haben wir - nachdem wir sowohl die Normalverteilungsannahme als auch die Homoskedastizitätsannahme gepüft haben - einen $t$-Test durchgeführt. Der Gruppenunterschied ist nicht bedeutsam ($t_{df = 164} = -0.273$, $p = 0.785$) - wir behalten die Nullhypothese also bei. Klinisch und nicht klinisch interessierte Studierende unterscheiden sich im Testwert für Verträglichkeit nicht bedeutsam.
Medianvergleich
Wir widmen uns nun der 2. Fragestellung. Dazu prüfen wir, ob klinisch interessierte Studierende in ihrer Lebenszufriedenheit im Mittel höhere Werte erreichen als Studierende, die sich primär für andere Inhalte interessieren. Hierbei handelt es sich also um eine gerichtete Hypothese. Außerdem wissen wir, dass die Lebenszufriedenheit in westlichen Ländern in der Population meist sehr schief verteilt ist. Statt also die Mittelwerte zu vergleichen, wollen wir in diesem Fall lieber die Mediane betrachten, um ein Maß der zentralen Tendenz zu nutzen, das sich besser für schiefe Verteilungen eignet. Also nutzen wir hier den Wilcoxon-Test.
Deskriptivstatistik
Wir beginnen damit uns, wie oben, erst einmal grafisch anzusehen, wie die Lebenszufriedenheit lz in beiden Gruppen aussieht. Dafür beginnen wir dieses mal mit einem Boxplot:
# Gruppierter Boxplot:
boxplot(fb23$lz ~ fb23$fach_klin,
xlab="Interesse", ylab="lz",
las=1, cex.lab=1.5,
main="Lebenszufriedenheit nach Interesse")
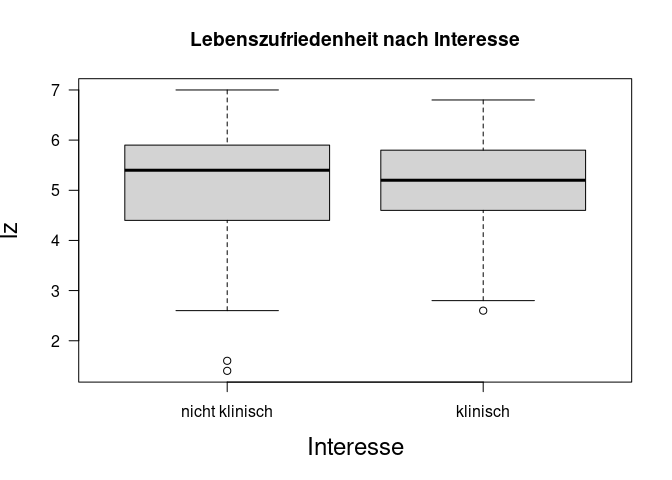
Hier können wir direkt (als dicke Linie eingezeichnet) die beiden Gruppenmediane sehen. Erst einmal sieht es so aus, als seien die beiden Mediane ein wenig unterschiedlich, wenn auch nicht drastisch. Für etwas mehr Detail, nutzen wir die Deskriptivstatistik aus der describeBy Funktion:
describeBy(fb23$lz, fb23$fach_klin) # beide Gruppen im Vergleich
##
## Descriptive statistics by group
## group: nicht klinisch
## vars n mean sd median trimmed mad min max range skew kurtosis se
## X1 1 83 5.11 1.13 5.4 5.19 1.19 1.4 7 5.6 -0.87 0.91 0.12
## --------------------------------------------------------------------------------------
## group: klinisch
## vars n mean sd median trimmed mad min max range skew kurtosis se
## X1 1 82 5.12 0.96 5.2 5.17 0.89 2.6 6.8 4.2 -0.49 -0.38 0.11
Auch hier sehen wir wieder, dass die Gruppe der nicht klinisch interessierten Studierenden einen etwas gößeren Median auf als die Gruppe der klinisch interessierten Studierenden.
Voraussetzungsprüfung
Wie schon der $t$-Test, hat auch der Wilcoxon-Test vier Voraussetzungen. Die ersten beiden bleiben gleich, nur die letzten beiden Unterscheiden sich leicht:
- zwei unabhängige Stichproben $\rightarrow$ ok
- die einzelnen Messwerte sind innerhalb der beiden Gruppen voneinander unabhängig (Messwert einer Vpn hat keinen Einfluss auf den Messwert einer anderen) $\rightarrow$ ok
- das untersuchte Merkmal ist stetig (mindestens singulär-ordinal skaliert)
- das Merkmal folgt in beiden Gruppen der gleichen Verteilung
Stetige Variablen haben theoretisch unendlich viele mögliche Ausprägungen. Dabei sind die Konsequenzen dieser Annahme jedoch nicht binär. Je mehr Ausprägungen eine Variable hat, desto besser funktioniert der Test. In unserem Fall ist die Variable lz ein Skalenwert, der sich als der Mittelwert von fünf Items mit jeweils sieben Antwortkategorien ergibt. Insgesamt sind das also 29 mögliche Abstufungen. Wir beschließen an dieser Stelle, dass 29 nah genug an unendlich dran ist, um von einer stetigen Variable zu sprechen.
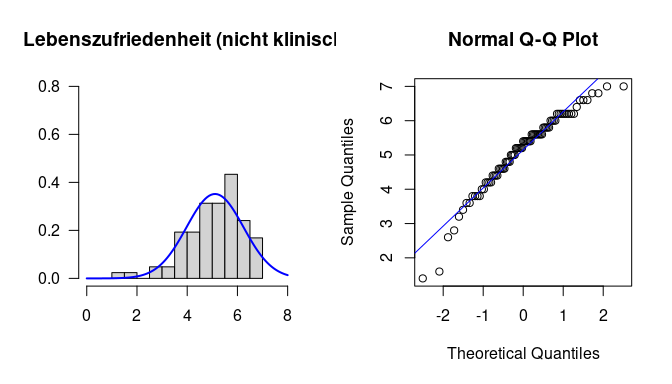
Um die Verteilung in beiden Gruppen zu untersuchen, gucken wir uns wieder die Histogramme und die QQ-Plots an:
# Gruppe 1 (nicht klinisch)
par(mfrow=c(1,2))
lz_nichtKlinisch <- fb23[(fb23$fach_klin=="nicht klinisch"), "lz"]
hist(lz_nichtKlinisch, xlim=c(0,8), ylim=c(0,.8), main="Lebenszufriedenheit (nicht klinisch)", xlab="", ylab="", las=1, probability=T)
curve(dnorm(x, mean=mean(lz_nichtKlinisch, na.rm=T), sd=sd(lz_nichtKlinisch, na.rm=T)), col="blue", lwd=2, add=T)
qqnorm(lz_nichtKlinisch)
qqline(lz_nichtKlinisch, col="blue")
# Gruppe 2 (klinisch)
par(mfrow=c(1,2))
lz_klinisch <- fb23[(fb23$fach_klin=="klinisch"), "lz"]
hist(lz_klinisch, xlim=c(0,8), ylim=c(0,.8), main="Lebenszufriedenheit (klinisch)", xlab="", ylab="", las=1, probability=T)
curve(dnorm(x, mean=mean(lz_klinisch, na.rm=T), sd=sd(lz_klinisch, na.rm=T)), col="blue", lwd=2, add=T)
qqnorm(lz_klinisch)
qqline(lz_klinisch, col="blue")
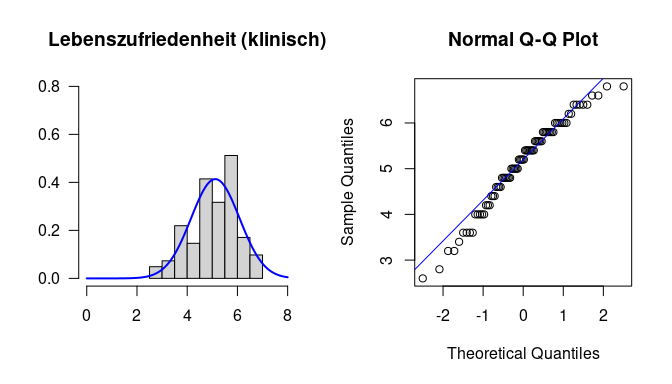 Wir sehen hier die vermutete Schiefe der Verteilung in beiden Gruppen, auch wenn diese sehr leicht ausfällt. Es sei an dieser Stelle gesagt, dass unter diesen Voraussetzungen ein $t$-Test auch funktionieren würde, weil die beiden Verteilungen nicht dramatisch von der Normalität abweichen und die beiden Stichproben jeweils relativ groß sind. Dennoch *wollen* wir hier den Wilcoxon-Test nutzen, weil wir nicht Mittelwerte, sondern Mediane vergleichen möchten. Die Verteilungen sehen in den beiden Gruppen sehr ähnlich aus. Wie man ganze Verteilungen auf Gleichheit prüft, werden wir im kommenden Semester noch näher betrachten, aber wenn zwei Verteilungen Gleich sind, impliziert dies auch, dass die Streuung in beiden Populationen gleich sein muss. Um diese Annahme zu prüfen, können wir auch hier den Levene-Test nutzen:
Wir sehen hier die vermutete Schiefe der Verteilung in beiden Gruppen, auch wenn diese sehr leicht ausfällt. Es sei an dieser Stelle gesagt, dass unter diesen Voraussetzungen ein $t$-Test auch funktionieren würde, weil die beiden Verteilungen nicht dramatisch von der Normalität abweichen und die beiden Stichproben jeweils relativ groß sind. Dennoch *wollen* wir hier den Wilcoxon-Test nutzen, weil wir nicht Mittelwerte, sondern Mediane vergleichen möchten. Die Verteilungen sehen in den beiden Gruppen sehr ähnlich aus. Wie man ganze Verteilungen auf Gleichheit prüft, werden wir im kommenden Semester noch näher betrachten, aber wenn zwei Verteilungen Gleich sind, impliziert dies auch, dass die Streuung in beiden Populationen gleich sein muss. Um diese Annahme zu prüfen, können wir auch hier den Levene-Test nutzen:leveneTest(fb23$lz ~ fb23$fach_klin)
## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 1 0.822 0.3659
## 163
Auch hier sehen wir ein nicht bedeutsames Ergebnis ($F_{1, 163} = 0.82$, $p = 0.366$), sodass wir die Nullhypothese beibehalten und die Annahme der Homoskedastizität nicht verwerfen müssen.
Inferenzstatistik mit dem Wilcoxon-Test
Zur Erinnerung, hier die Fragestellung, die wir untersuchen wollten:
Erreichen klinisch interessierte im Gegensatz zu nicht klinisch interessierten Studierenden in der Lebenszufriedenheit im Mittel höhere Werte?
Wir müssen aus dieser Fragestellung also eine gerichtete Hypothese ableiten:
- $H_0$: Klinisch interessierte Studierende erreichen im Mittel niedrigere oder genauso hohe Werte der Lebenszufriedenheit wie nicht klinisch interessierte Studierende.
- $H_1$: Klinisch interessierte Studierende erreichen im Mittel höhere Werrte der Lebenszufriedenheit als nicht klinisch interessierte Studierende.
Wieder etwas formaler ausgedrückt:
- $H_0$: $\eta_\text{klinisch} \leq \eta_\text{nicht klinisch}$ bzw. $\eta_\text{klinisch} - \eta_\text{nicht klinisch} \leq 0$
- $H_1$: $\eta_\text{klinisch} \gt \eta_\text{nicht klinisch}$ bzw. $\eta_\text{klinisch} - \eta_\text{nicht klinisch} \gt 0$
Wie so häufig, nehmen wir auch hier ein $\alpha$-Fehlerniveau von 5% an.
Die Funktion wilcox.test() nimmt im Grunde die gleichen Argumente entgegen wie die Funktion t.test(). Damit wir die gerichtete Hypothese in die richtige Richtung aufstellen können, müssen wir wissen, welches das erste Level des Faktors ist:
levels(fb23$fach_klin) # wichtig zu wissen: die erste der beiden Faktorstufen ist "nicht klinisch"
## [1] "nicht klinisch" "klinisch"
Da die erste Faktorstufe “nicht klinisch” ist, wissen wir, dass die gerichtete Hypothese “<” lauten muss. In R wird mit dem Argument alternative immer Bezug genommen auf die Formulierung der Alternativhypothese. In unseren Fall ist diese, dass die erste Gruppe (nicht klinisch) einen niedrigeren Median hat, als die zweite Gruppe (klinisch), also müssen wir alternative = 'less' angeben:
wilcox.test(fb23$lz ~ fb23$fach_klin, # abhängige Variable ~ unabhängige Variable
paired = FALSE, # Stichproben sind unabhängig
alternative = "less", # einseitige Testung
conf.level = .95) # alpha = .05
##
## Wilcoxon rank sum test with continuity correction
##
## data: fb23$lz by fb23$fach_klin
## W = 3467, p-value = 0.5834
## alternative hypothesis: true location shift is less than 0
Per Voreinstellung wird in R der exakte $p$-Wert bestimmt, wenn die Stichprobe insgesamt weniger als 50 Personen umfasst und keine Rangbindungen vorliegen. Bei größeren Stichproben folgt die Rangsumme aufgrund des zentralen Grenzwertsatzes ausreichend gut der Normalverteilung und es wird ein $z$-Test der Rangsumme durchgeführt. Weil die Rangsumme allerdings nur ganze Zahlen annehmen kann ist diese Approximation ein wenig ungenau (insbesondere dann, wenn die Stichprobe noch relativ klein ist). Per Voreinstellung wird daher in R eine Kontinuitätskorrektur durchgeführt (mit dem Argument correct = TRUE), sodass Sie nicht den gleichen Wert erhalten, den Sie bekommen, wenn Sie den Test händisch durchführen würden. Wenn Sie diese Korrektur ausschalten (correct = FALSE) erhalten Sie den gleichen Wert.
Ergebnisinterpretation
Wir haben untersucht, ob klinisch interessierte Studierende im Mittel höhere Werte in der Lebenszufriedenheit erreichen als nicht klinisch interessierte Studierende. Deskriptiv besteht kaum ein Unterschied: Beide Gruppen weisen einen ähnlichen Median auf: $Md_\text{nicht klinisch}$ = 5.4 und $Md_\text{klinisch}$ = 5.2. Zur Überprüfung der Hypothese wurde ein Wilcoxon-Test durchgeführt. Der Gruppenunterschied ist nicht statistisch bedeutsam ($W = 3467$, $p = 0.583$). Somit wird die Nullhypothese beibehalten: Klinisch interessierte Studierende erreichen keine höheren Werte in der Lebenszufriedenheit als nicht klinisch interessierte Studierende.
In diesem Beitrag haben wir gesehen, wie wir die zentrale Tendenz (Mittelwert oder Median) bei zwei unabhängigen Stichproben untersuchen können. Im nächsten Beitrag sehen wir dann, wie das Ganze funktioniert, wenn wir zwei Gruppen betrachten, zwischen denen eine Abhängigkeit besteht (z.B. weil wir wiederholt die gleichen Personen untersucht haben). Im Anhang finden Sie außerdem noch einen Test, mit dem Sie untersuchen können, ob die Häufigkeitsverteilungen in zwei unabhängigen Stichproben sich unterscheidet. Dieser Test bietet also die Möglichkeit auch nominalskalierte abhängige Variablen über zwei Gruppen zu vergleichen.
Appendix
Vierfelder-$\chi^2$-Test
Gehen wir nun zur 3. Fragestellung über. Wir verwenden wieder den Datensatz fb23.
Zusätzlich zur Gruppenvariable ist in diesem Beispiel auch die abhängige Variable nominalskaliert. Um Fragen wie diese zu beantworten, werden daher die Populationswahrscheinlichkeiten (job: ja [ja] vs. nein [nein]) zwischen den beiden Gruppen (ort: Frankfurt [FFM] vs. außerhalb [andere]) miteinander verglichen. Diese Prüfung erfolgt mithilfe des $\chi^2$-Tests.
Datenaufbereitung
Zunächst müssen wir den ort und den job als Faktor abspeichern und entsprechende Labels vergeben. Damit wir hier keine Probleme bekommen, müssen wir zunächst prüfen, ob die Variablen ein factor sind:
is.factor(fb23$ort)
## [1] FALSE
is.factor(fb23$job)
## [1] FALSE
Dies ist bei beiden nicht der Fall, weswegen wir hier die Variable als Faktor ablegen können. Wir verwenden die Labels, die wir oben bereits in Klammern geschrieben haben.
# Achtung, nur einmal durchführen (ansonsten Datensatz neu einladen und Code erneut durchlaufen lassen!)
fb23$ort <- factor(fb23$ort, levels=c(1,2), labels=c("FFM", "anderer"))
fb23$job <- factor(fb23$job, levels=c(1,2), labels=c("nein", "ja"))
Deskriptivstatistik und Voraussetzungsprüfung
Wir beginnen wieder damit, uns Deskriptivstatistiken anzusehen und die Voraussetzungen des $\chi^2$-Tests zu prüfen. Diese sind
- Die einzelnen Beobachtungen sind voneinander unabhängig $\rightarrow$ ok (durch das Studiendesign anzunehmen)
- Jede Person lässt sich eindeutig einer Kategorie bzw. Merkmalskombination zuordnen $\rightarrow$ ok (durch das Studiendesign anzunehmen)
- Zellbesetzung für alle $n_{ij}$ > 5 $\rightarrow$ Prüfung anhand von Häufigkeitstabelle
Weil es sich beim $\chi^2$-Test um einen parametrischen Test handelt, folgt die Teststastistik genau genommen erst bei ausreichend großen Stichproben tatsächlich der behaupteten Verteilung. Für den Vierfelder-Test reicht es eigentlich schon aus, dass jede Zelle mit mindestens 5 Fällen besetzt ist:
tab <- table(fb23$ort, fb23$job)
tab
##
## nein ja
## FFM 61 54
## anderer 35 25
Hier sollten uns aus einer zu geringen Häufigkeit in einer Zelle der Häufigkeitstabelle also keine Probleme entstehen.
Durchführung des $\chi^2$-Tests
Hypothesenpaar (inhaltlich):
- $H_0$: Studierende mit Wohnort in Uninähe haben mit gleicher Wahrscheinlichkeit einen Nebenjob wie Studierende, deren Wohnort außerhalb von Frankfurt liegt.
- $H_1$: Studierende mit Wohnort in Uninähe haben mit einer höheren oder niedrigeren Wahrscheinlichkeit einen Nebenjob als Studierende, deren Wohnort außerhalb von Frankfurt liegt.
Hypothesenpaar (statistisch):
- $H_0$: $\pi_{ij} = \pi_{i\bullet} \cdot \pi_{\bullet j}$
- $H_1$: $\pi_{ij} \neq \pi_{i\bullet} \cdot \pi_{\bullet j}$
wobei $\pi_{ij}$ die Wahrscheinlichkeit, in Zelle $ij$ zu landen, ist. Betrachten wir die Häufigkeitstabelle, dann entspricht dies der Wahrscheinlichkeit der Ausprägung der $i$-ten Zeile (Variable: Wohnortnähe) und der $j$-ten Spalte (Variable: Nebenjob). $\pi_{i\bullet}$ beschreibt die Wahrscheinlichkeit, die Ausprägung der $i$-ten Zeile (Variable: Wohnortnähe) zu haben. $\pi_{\bullet j}$ beschreibt die Wahrscheinlichkeit, die Ausprägung der der $j$-ten Spalte (Variable: Nebenjob) zu haben. Die beiden betrachteten Variablen sind voneinander unabhängig, wenn die Wahrscheinlichkeit für das Auftreten von Y (Nebenjob) nicht davon abhängt, welches X (Wohnort) vorliegt. Ist dies der Fall, lässt sich die Wahrscheinlichkeit für das Auftreten der Kombination der Merkmalsausprägungen in das Produkt der Einzelwahrscheinlichkeiten zerlegen. Bspw. sollte sich die Wahrscheinlichkeit in Frankfurt zu wohnen und keinen Nebenjob zu haben, ausdrücken lassen (gleich sein) mit der Wahrscheinlichkeit in Frankfurt zu wohnen multipliziert mit der Wahrscheinlichkeit keinen Nebenjob zu haben: $\pi_\text{FFM,nein}=\pi_{\text{FFM},\bullet}\cdot\pi_{\bullet,\text{nein}}$ (unter $H_0$).
Für jede Zelle lassen sich die unter Gültigkeit der Nullhypothese erwarteten Häufigkeiten $e_{ij}$ bestimmen (hier sind $n_{i\bullet}$ und $n_{\bullet j}$ die absoluten Häufigkeiten), die zu den Wahrscheinlichkeiten aus dem Hypothesenpaar gehören:
$$e_{ij} = \frac{n_{i\bullet} \cdot n_{\bullet j}}{n}$$
$n_{i\bullet}$ und $n_{\bullet j}$ lassen sich über die Randsummen bestimmen (sie symbolisieren eigentlich: $n_{i\bullet}=n\hat{\pi}_{i\bullet}$, mit $n$ als Stichprobengröße). Diese hängen wir unseren Daten an. Anschließend erstellen wir einen neuen Datensatz und fügen alle erwarteten Häufigkeiten dort ein.
tab_mar <- addmargins(tab) # Randsummen zu Tabelle hinzufügen
tab_mar
##
## nein ja Sum
## FFM 61 54 115
## anderer 35 25 60
## Sum 96 79 175
expected <- data.frame(nein=c((tab_mar[1,3]*tab_mar[3,1])/tab_mar[3,3],
(tab_mar[2,3]*tab_mar[3,1])/tab_mar[3,3]),
ja=c((tab_mar[1,3]*tab_mar[3,2])/tab_mar[3,3],
(tab_mar[2,3]*tab_mar[3,2])/tab_mar[3,3]))
expected
## nein ja
## 1 63.08571 51.91429
## 2 32.91429 27.08571
Bspw. für die Kombination (FFM, nein) ergibt sich eine erwartete Häufigkeit von 63.09, welcher eine beobachtete Häufigkeit von 61 gegenüber steht. Mit dem $\chi^2$-Test können wir nun bestimmen, ob diese Unterschiede statistisch bedeutsam groß sind.
Um die Prüfgröße $\chi^2$ zu berechnen, können wir folgende Formel nutzen: $$\chi^2 = \sum_{i=1}^{2}{ \sum_{j=1}^{2}{ \frac{(n_{ij}-e_{ij})^2} {e_{ij}}}}$$
chi_quadrat_Wert <- (tab[1,1]-expected[1,1])^2/expected[1,1]+
(tab[1,2]-expected[1,2])^2/expected[1,2]+
(tab[2,1]-expected[2,1])^2/expected[2,1]+
(tab[2,2]-expected[2,2])^2/expected[2,2]
chi_quadrat_Wert
## [1] 0.4455294
Die Freiheitsgrade berechnen sich aus der Anzahl der untersuchten Kategorien: $df = (p - 1) \cdot (k - 1)$. Hier, im Fall des Vierfelder-$\chi^2$-Tests also mit $df = 1$, wobei
- p: Anzahl Kategorien Variable “ort” = 2
- k: Anzahl Kategorien Variable “job” = 2
Zur Bestimmung des kritischen Wertes und des $p$-Wertes ziehen wir die jeweiligen Funktionen der $\chi^2$ -Verteilung heran:
qchisq(.95, 1) # kritischer Wert
## [1] 3.841459
pchisq(chi_quadrat_Wert, 1, lower.tail = FALSE) # p-Wert
## [1] 0.5044657
Insgesamt ergibt sich damit.
- df = 1
- $\chi^2_{krit}$ = 3.84
- $\chi^2_{emp}$ = 0.446
- $\chi^2_{emp} < \chi^2_{krit}$ $\rightarrow H_0$ wird beibehalten
- $p$ = 0.504
- $p$-Wert > $\alpha$-Fehlerniveau $\rightarrow H_0$ wird beibehalten
Daraus lässt sich zusammenfassen: Die Wohnnähe zur Uni hängt nicht damit zusammen, ob ein Nebenjob ausgeübt wird $\chi^2$(df=1)=0.446, $p$ = 0.504.
Im Normalfall übernimmt die Funktion chisq.test() die Arbeit für uns, wenn wir ihr einfach eine 4-Feldertabelle übergeben. Als Argumente brauchen wir hauptsächlich das Objekt mit den Häufigkeiten. Weiterhin müssen wir das Argument correct mit FALSE angeben. Ansonsten würde standardmäßig die Kontinuitätskorrektur nach Yates durchgeführt werden. Diese ist als Alternative gedacht, wenn nicht der $\chi^2$-Verteilung gefolgt wird. Ein Indiz dafür sind bspw. sehr kleine Zahlen in manchen Kombinationen der Faktoren. Da wir hier in jeder Zelle einen größeren Wert hatten, brauchen wir diese Korrektur nicht.
chisq.test(tab, # Kreuztabelle
correct=F) # keine Kontinuinitätskorrektur nach Yates
##
## Pearson's Chi-squared test
##
## data: tab
## X-squared = 0.44553, df = 1, p-value = 0.5045
Das Ergebnis unterscheidet sich natürlich nicht zu unserer händischen Berechnung. Die Wohnnähe zur Uni hängt nicht damit zusammen, ob ein Nebenjob ausgeübt wird.
Effektstärken
Auch für den $\chi^2$-Test gibt es die Möglichkeit zur Einordnung der Effektstärke - in diesem Fall sind es sogar zwei verschiedene Varianten:
Yules Q
Dieses berechnet sich als
$$Q=\frac{n_{11}n_{22}-n_{12}n_{21}}{n_{11}n_{22}+n_{12}n_{21}},$$
welches einen Wertebereich von [-1,1] aufweist und analog zur Korrelation interpretiert werden kann. 1 steht in diesem Fall für einen perfekten positiven Zusammenhang (dazu in der entsprechenden Sitzung mehr).
In R sieht das so aus:
effekt_YulesQ <- (tab[1,1]*tab[2,2]-tab[1,2]*tab[2,1])/
(tab[1,1]*tab[2,2]+tab[1,2]*tab[2,1])
effekt_YulesQ
## [1] -0.1068814
Phi ($\phi$)
Auch kann man $\phi$ bestimmen:
$$\phi = \frac{n_{11}n_{22}-n_{12}n_{21}}{\sqrt{(n_{11}+n_{12})(n_{11}+n_{21})(n_{12}+n_{22})(n_{21}+n_{22})}}$$ welches einen Wertebereich von [-1,1] aufweist und analog zur Korrelation interpretiert werden kann. 1 steht in diesem Fall für einen perfekten positiven Zusammenhang (dazu in der entsprechenden Sitzung mehr).
In R sieht das so aus:
effekt_phi <- (tab[1,1]*tab[2,2]-tab[1,2]*tab[2,1])/
sqrt((tab[1,1]+tab[1,2])*(tab[1,1]+tab[2,1])*(tab[1,2]+tab[2,2])*(tab[2,1]+tab[2,2]))
effekt_phi
## [1] -0.05045674
Das Ganze lässt sich auch mit dem psych und der darin enthaltenen Funktion phi() umsetzen:
# alternativ mit psych Paket
library(psych)
phi(tab, digits = 8)
## [1] -0.05045674
Yule(tab)
## [1] -0.1068814
# Äquivalentes Ergebnis mittels Pearson-Korrelation (kommt in den nächsten Sitzungen)
# (dichotome Variablen)
ort_num <- as.numeric(fb23$ort)
job_num <- as.numeric(fb23$job)
cor(ort_num, job_num, use="pairwise")
## [1] -0.05045674
Cohen (1988) hat folgende Konventionen zur Beurteilung der Effektstärke $\phi$ vorgeschlagen, die man heranziehen kann, um den Effekt “bei kompletter Ahnungslosigkeit” einschätzen zu können (wissen wir mehr über den Sachverhalt, so sollten Effektstärken lieber im Bezug zu anderen Studienergebnissen interpretiert werden):
| phi | Interpretation |
|---|---|
| ~ .1 | kleiner Effekt |
| ~ .3 | mittlerer Effekt |
| ~ .5 | großer Effekt |
Der Wert für den Zusammenhang der beiden Variablen ist also bei völliger Ahnungslosigkeit als klein einzuschätzen.
Ergebnisinterpretation
Es wurde untersucht, ob Studierende mit Wohnort in Uninähe (also in Frankfurt) mit gleicher Wahrscheinlichkeit einen Nebenjob haben wie Studierende, deren Wohnort außerhalb von Frankfurt liegt. Zur Beantwortung der Fragestellung wurde ein Vierfelder-Chi-Quadrat-Test für unabhängige Stichproben berechnet. Der Zusammenhang zwischen Wohnort und Nebenjob ist nicht signifikant ($\chi^2$(1) = 0.446, p = 0.504), somit wird die Nullhypothese beibehalten. Der Effekt ist von vernachlässigbarer Stärke ($\phi$ = -0.05). Studierende mit Wohnort in Uninähe haben mit gleicher Wahrscheinlichkeit einen Nebenjob wie Studierende, deren Wohnort außerhalb von Frankfurt liegt.
](/media/header/writing_math.jpg)