Einleitung und Wiederholung
Einleitung
Im Verlauf des Seminars Forschungsmethoden und Evaluation I soll neben der Einführung in die Theorie und Hintergründe multivariater Verfahren auch eine Einführung in deren Umsetzung gegeben werden, sodass Sie in der Lage sind, diese Verfahren in Ihrem zukünftigen akademischen und beruflichen Werdegang zu benutzen. R ist eine freie Software, die vor allem für (statistische) Datenanalysen verwendet wird. Bevor wir uns die Regressionsanalyse in R ansehen wollen, sollten Sie sich etwas mit R vertraut gemacht sowie die nötige Software (R als Programmiersprache und R-Studio als schöneres Interface) installiert haben. Hierzu eignet sich hervorragend der ebenfalls auf Pandar zu findende R-Crash Kurs. Dort gibt es die Intro und Installationsanweisung sowohl in deutscher als auch in englischer Sprache. Wir gehen davon aus, dass Sie diese durchgearbeitetet haben und schauen uns als Einführung zunächst ein paar zentrale Befehle für das Dateneinlesen und -verarbeiten noch einmal an.
Nach dem Ende der Übung finden Sie in dem zu dieser Veranstaltung gehörenden OLAT Kurs Aufgaben (ein Quiz), die die Inhalte dieser Sitzung ergänzen.
Daten einlesen und verarbeiten
Falls Sie an der Goethe-Universität studiert haben, kennen Sie den Datensatz, den wir in dieser Sitzung untersuchen wollen, vielleicht aus dem Bachelorstudium: Eine Stichprobe von 100 Schülerinnen und Schülern hat einen Lese- und einen Mathematiktest, sowie zusätzlich einen allgemeinen Intelligenztest, bearbeitet. Im Datensatz enthalten ist zudem das Geschlecht (female, 0=m, 1=w). Sie können den Datensatz “Schulleistungen.rda” hier herunterladen.
Daten laden
Der Datensatz ist im R-internen “.rda” Format abgespeichert - einem datensatzspezifischen Dateiformat. Wir können diesen Datensatz einfach mit der load Funktion laden. Wir müssen R nur mitteilen, wo der Datensatz liegt und è vola, er wird uns zur Verfügung gestellt. Liegt der Datensatz bspw. auf dem Desktop, so müssen wir den Dateipfad dorthin legen und können dann den Datensatz laden (wir gehen hier davon aus, dass Ihr PC “Musterfrau” heißt):
load("C:/Users/Musterfrau/Desktop/Schulleistungen.rda")
Nun sollte in R-Studio oben rechts in dem Fenster unter der Rubrik “Data” unser Datensatz mit dem Namen “Schulleistungen” erscheinen.
Bei Dateipfaden ist darauf zu achten, dass bei Linux
oder Mac OS Rechnern
immer Front-Slashes ("/") zum Anzeigen von Hierarchien zwischen Ordnern verwendet werden, während auf Windows Rechnern
im System aber bei Dateipfaden mit Back-Slashes gearbeitet wird ("\"). R nutzt auf Windows Rechnern
ebenfalls Front-Slashes ("/"). Das bedeutet, dass, wenn wir auf Windows Rechnern
den Dateipfad aus dem Explorer kopieren, wir die Slashes “umdrehen” müssen.
Genauso sind Sie in der Lage, den Datensatz direkt aus dem Internet zu laden. Hierzu brauchen Sie nur die URL und müssen R sagen, dass es sich bei dieser um eine URL handelt, indem Sie die Funktion url auf den Link anwenden. Der funktionierende Befehl sieht so aus (wobei die URL in Anführungszeichen geschrieben werden muss):
load(url("https://pandar.netlify.app/daten/Schulleistungen.rda"))
An diesem Link erkennen wir, dass Websiten im Grunde auch nur schön dargestellte Ordnerstrukturen sind. So liegt auf der Pandar-Seite, die auf netlify.app gehostet wird, ein Ordner namens daten, in welchem wiederum das Schulleistungen.rda liegt.
Ein Überblick über die Daten
Wir können uns die ersten (6) Zeilen des Datensatzes mit der Funktion head ansehen. Dazu müssen wir diese Funktion auf den Datensatz (das Objekt) Schulleistungen anwenden:
head(Schulleistungen)
## female IQ reading math
## 1 1 81.77950 449.5884 451.9832
## 2 1 106.75898 544.8495 589.6540
## 3 0 99.14033 331.3466 509.3267
## 4 1 111.91499 531.5384 560.4300
## 5 1 116.12682 604.3759 659.4524
## 6 0 106.14127 308.7457 602.8577
Wir erkennen die 4 Spalten mit dem Geschlecht, dem IQ, der Lese- und der Mathematikleistung. Außerdem sehen wir, dass die Variablen mit den Namen IQ, reading und math einige Dezimalstellen aufweisen, was daran liegt, dass diese Daten simuliert wurden (allerdings echten Daten nachempfunden sind). Da es sich bei unserem Datensatz um ein Objekt vom Typ data.frame handelt, können wir die Variablennamen des Datensatzes außerdem mit der names Funktion abfragen. Weitere interessante Funktionen sind die nrow und ncol Funktion, die, wie Sie sich sicher schon gedacht haben, die Zeilen- und die Spaltenanzahl wiedergeben (wir könnten auch dim verwenden, was diese beiden Informationen auf einmal ausgibt).
# Namen der Variablen abfragen
names(Schulleistungen)
## [1] "female" "IQ" "reading" "math"
# Anzahl der Zeilen
nrow(Schulleistungen)
## [1] 100
# Anzahl der Spalten
ncol(Schulleistungen)
## [1] 4
# Anzahl der Zeilen und Spalten kombiniert
dim(Schulleistungen)
## [1] 100 4
# Struktur des Datensatzes - Informationen zu Variablentypen
str(Schulleistungen)
## 'data.frame': 100 obs. of 4 variables:
## $ female : num 1 1 0 1 1 0 1 1 1 1 ...
## $ IQ : num 81.8 106.8 99.1 111.9 116.1 ...
## $ reading: num 450 545 331 532 604 ...
## $ math : num 452 590 509 560 659 ...
Wir entnehmen dem Output, dass die Variablen im Datensatz female, IQ, reading und math heißen, dass insgesamt $n = 100$ Schulkinder zu 4 Merkmalen befragt/untersucht wurden und dass alle diese Variablen “numerisch”, also als Zahlen, vorliegen. Eine einzelne Variable eines Datensatzes (vom Typ data.frame) können wir ansprechen, indem wir hinter den Datensatz $ schreiben und anschließend den Variablennamen. Zugriff auf den IQ der Kinder erhalten wir mit:
Schulleistungen$IQ
## [1] 81.77950 106.75898 99.14033 111.91499 116.12682 106.14127 85.44854 93.24323
## [9] 135.19738 89.90152 92.72073 115.90123 114.54088 83.28294 126.41670 107.20436
## [17] 90.03418 98.34044 117.06874 115.55140 68.11351 125.64776 93.34804 106.93651
## [25] 98.78466 78.93267 113.05378 92.86905 86.44483 70.17249 111.44613 93.78654
## [33] 87.54754 87.01957 69.32581 92.85801 70.56712 74.17486 105.61195 110.63901
## [41] 91.54624 105.73141 125.26215 101.14873 111.09582 79.99545 84.45429 84.50532
## [49] 96.60821 103.90556 81.03395 126.12813 89.47650 80.78064 106.48847 103.58060
## [57] 84.88878 115.90930 97.28407 91.60586 121.77877 110.26187 100.32137 112.65157
## [65] 122.84032 96.45124 75.48471 91.27550 111.85776 92.72890 76.84326 92.93814
## [73] 103.25579 81.15462 92.27190 106.40950 96.70280 104.06385 107.98499 60.76781
## [81] 94.55947 103.55973 101.83276 113.06302 76.56824 97.56684 104.28662 106.08550
## [89] 120.97759 82.65717 108.41181 103.38963 100.59534 122.79791 97.91853 92.96729
## [97] 77.51862 105.01989 54.05485 106.12641
Tipp: In R-Studio können Sie sich Ihren Umgang mit der Software in vielen Dingen vereinfachen, indem Sie die automatische Vervollständigung der Software nutzen. Dies tun Sie, indem Sie bspw. Schulleistungen$ tippen und dann den Tabulator [oder “Strg” + “Leertaste” auf Windows
oder Linux Rechner
oder “Control” + “Leertaste” auf Mac OS Rechnern
] auf Ihrer Tastatur drücken. Ihnen werden dann Vorschläge für mögliche Argumente aufgezeigt. Das gleiche funktioniert auch in Funktionen. Hier müssen Sie zunächst den Funktionsnamen schreiben und die Klammern öffnen. Mit dem Tabulator erhalten Sie anschließend Vorschläge für mögliche Argumente, die Sie der Funktion übergeben können. Schauen Sie sich dies doch einmal an! Dies funktioniert übrigens auch für das Vervollständigen von Dateipfaden. Hierbei muss allerdings darauf geachtet werden, dass diese in Anführungsstrichen geschrieben werden und Sie müssen beachten, wo ihr aktuelles Working-Directory liegt. Sie können allerdings auch den vollständigen Pfad eingeben, indem Sie auf Windows PCs
mit “C:/Users/” und auf Mac OS Rechnern
mit “/Users/” beginnen und dann den Tabulator drücken und den jeweilig richtigen Ordner auswählen, bis Sie an Ihrer Zieldatei angekommen sind!
Einfache Deskriptivstatistiken
Bevor wir jetzt direkt zu komplexeren Analysen springen, wollen wir uns noch schnell mit dem Bestimmen einfacher deskriptivstatistischer Größen vertraut machen. Mit der Funktion sum können wir die Summe und mit mean können wir den Mittelwert einer Variable bestimmen. Eine Schätzung für die Populationsvarianz erhalten wir mit var. Die zugehörige Populationsschätzung für die Standardabweichung, Sie vermuten es vielleicht, erhalten wir mit sd. Es handelt sich hierbei um Populations- und nicht um Stichprobenschätzungen, da in diesen Funktionen der Vorfaktor $\frac{1}{n-1}$ genutzt wird, um einen unverzerrten Schätzer für eben die Variation in der Population zu erhalten (für mehr Informationen siehe bspw. Eid, Gollwitzer und Schmitt, 2017, S. 162-163 in Kapitel 6.4.4 und S. 246-247 in Kapitel 8.5.1 oder Agresti, & Finlay, 2013, Kapitel 3.2 und folgend).
# Summe
sum(Schulleistungen$IQ)
## [1] 9813.425
# Mittelwert
mean(Schulleistungen$IQ)
## [1] 98.13425
1/100*sum(Schulleistungen$IQ) # auch der Mittelwert
## [1] 98.13425
# Varianz
var(Schulleistungen$IQ)
## [1] 248.1075
# SD
sd(Schulleistungen$IQ)
## [1] 15.75143
sqrt(var(Schulleistungen$IQ)) # die Wurzel aus der Varianz ist die SD, hier: sqrt() ist die Wurzel Funktion
## [1] 15.75143
Mit summary können wir uns die Zusammenfassung einer Variable ansehen.
summary(Schulleistungen$IQ)
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 54.05 87.42 98.56 98.13 108.09 135.20
Als Ausgabe erhalten wir den kleinsten Wert 54.05 (das Minimum, welches hier bspw. auf 2 Nachkommastellen gerundet ist; Originalwert = 54.0548457), das erste Quartil 87.42, also den 25.-Prozentrang (25% PR), den Median 98.56 (2. Quartil; 50% PR), den Mittelwert 98.13, das 3. Quartil 108.09 (75% PR) und den größten Wert 135.2 (Maximum). Wenden wir diese Funktion auf ein R-Objekt an, so gibt uns diese eine Zusammenfassung dieses Objekts aus, was häufig auch einfach die Zusammenfassung einer Analyse ist. Dies schauen wir uns später nochmals genauer an.
Als letztes gucken wir uns noch den Befehl colMeans an, welcher Mittelwerte eines Datensatzes über die Spalten (also pro Variable über alle Personen) bestimmt. Somit lassen sich ganz einfach für alle Variablen eines Datensatzes auf einmal die Mittelwerte bestimmen (rowMeans bestimmt, wie Sie sich wahrscheinlich denken, die Mittelwerte pro Zeile, also die Mittelwerte über alle Variablen pro Person):
colMeans(Schulleistungen)
## female IQ reading math
## 0.54000 98.13425 496.06605 561.46446
Da female 0-1 kodiert ist und 1 für Mädchen steht, bedeutet die hier beobachtete Zahl von .54, dass gerade ca. 54% der Proband*innen weiblich waren.
Der $t$-Test
Ein sehr einfacher statistischer Test, ist der $t$-Test (für eine Wiederholung siehe bspw. Eid, et al., 2017, Kapitel 11.1 oder Agresti, & Finlay, 2013, Kapitel 6.2 und folgend). Mit Hilfe dieses Tests soll untersucht werden, ob die Mittelwerte in zwei Gruppen gleich sind. Dazu brauchen wir drei wichtige Annahmen:
- die Beobachtungen stammen aus einer independent and identically distributed (i.i.d., deutsch: unabhängig und identisch verteilt) Population (dies bedeutet, dass alle Beobachtungen unabhängig sind und den gleichen Verteilungs- und Modellannahmen unterliegen),
- die Daten in beiden Gruppen sind normalverteilt mit
- gleicher Varianz (Varianzhomogenität).
Die 1. Annahme ist sehr kritisch und lässt sich leider nicht prüfen. Sie lässt sich aber durch das Studiendesign zumindest plausibilisieren. Für die anderen beiden Annahmen gibt es verschiedene Tests und deskriptive Verfahren, um ein Gefühl für ihr Vorliegen zu erhalten - diese schauen wir uns in der nächsten Sitzung zur Regression an. Unter diesen Annahmen stellen wir die folgenden Null-Hypothese auf - die Gleichheit der Mittelwerte in den beiden Gruppen: $$H_0: \mu_1=\mu_2$$
Diese Hypothese gilt nicht, wenn $\mu_1\neq\mu_2$. In diesem Fall gilt irgendeine Alternativhypothese ($H_1$) mit einer Mittelwertsdifferenz $d=\mu_1-\mu_2$, die nicht Null ist $(d\neq0)$. Wir sprechen hier von einer Alternativhypothese, da in der (frequentistischen) Statistik immer feste Werte für die Parameter in der Population angenommen werden müssen. Wenn die $H_0$ nicht gilt, so muss entsprechend auch ein fester Wert für die Differenz $d$ für die Mittelwerte angenommen werden.
Simulieren von Daten
Wir können in R auch Daten simulieren. Bspw. erzeugt der Befehl rnorm normalverteilte Zufallsvariablen. Für weitere Informationen siehe bspw. R-Verteilungen auf Wiki. Wir müssen diesem Befehl lediglich übergeben wie viele Replikationen wir wünschen und welchen Mittelwert und Standardabweichung die Zufallsvariablen haben sollen. Wir simulieren die Standardnormalverteilung $\mathcal{N}(0,1)$ und legen die generierte (realisierte) Zufallsvariable in einem Objekt mit dem Namen X ab, um später gezeigte Informationen wie den Mittelwert oder die Standardabweichung abrufen zu können - dies machen wir mit dem “Zuordnungspfeil” <- (zur Erinnerung: links davon steht der Name, den wir uns ausdenken; hier: X; rechts steht das zugeordnete Objekt). Um zufällige Prozesse über verschiedene Zeitpunkte hinweg konstant zu halten (damit es replizierbar ist und Sie dasselbe Ergebnis bekommen), wird zunächst ein Startpunkt für den Zufallsprozess mittels set.seed gesetzt. set.seed nimmt irgendeine ganzzahlige, positive Zahl entgegen.
set.seed(1234567) # für Replizierbarkeit (bei gleicher R Version, kommen Sie mit diesem Seed zum selben Ergebnis!)
X <- rnorm(n = 1000, mean = 0, sd = 1) # Standardnormalverteilung mit n = 1000
hist(X, breaks = 50) # breaks gibt die Anzahl der Balken vor
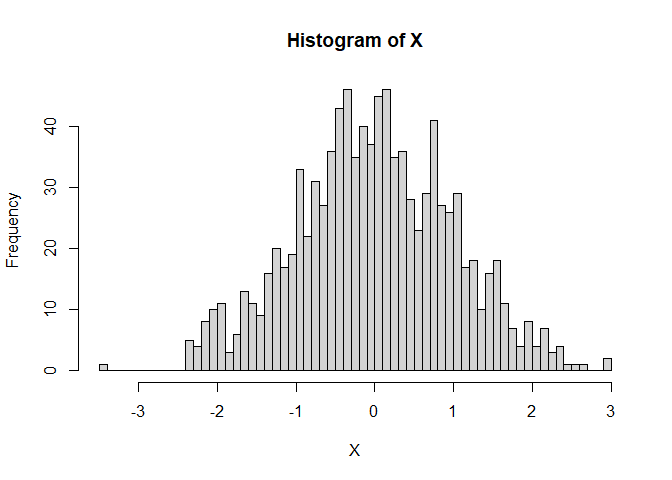
mean(X)
## [1] -0.002997332
sd(X)
## [1] 0.9956916
Das was in rnorm drin steckt, nämlich eine Normalverteilung mit Mittelwert 0 und Standardabweichung 1, ist gerade die Population, aus der wir eine Stichprobe gezogen haben. Natürlich liegen der geschätzte Mittelwert und die geschätze Standardabweichung der Stichprobe nicht genau auf den wahren Werten der Population, was am Sampling-Error liegt. Der Sampling-Error beschreibt die zufällige Schwankung, die durch das Ziehen der Stichprobe aus einer Population (Grundgesamtheit) entsteht.
Gehen wir nun her und simulieren noch eine zweite Stichprobe standardnormalverteilter Zufallsvariablen, so können wir diese beiden Stichproben mit Hilfe des $t$-Tests vergleichen. Da in beiden Gruppen ein Mittelwert von 0 vorliegt, ziehen wir hier einen $t$-Test heran im Fall der Null-Hypothese. Die Funktion in R hierzu heißt t.test (wir müssen hier var.equal = T wählen, da sonst eine Näherung verwendet wird, die unterschiedliche Varianzen ausgleichen soll, nämlich Welch’s Two Sample $t$-Test):
set.seed(2)
Y <- rnorm(n = 1000, mean = 0, sd = 1)
t.test(X, Y, var.equal = T)
##
## Two Sample t-test
##
## data: X and Y
## t = -1.4456, df = 1998, p-value = 0.1484
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -0.15317201 0.02317988
## sample estimates:
## mean of x mean of y
## -0.002997332 0.061998736
Der Output enthält folgende Informationen:
##
## Two Sample t-test
zeigt an, dass es sich um einen Zwei-Stichproben $t$-Test handelt.
## data: X and Y
## t = -1.4456, df = 1998, p-value = 0.1484
zeigt uns die Datengrundlage (X und Y), den $t$-Wert, die $df$ und den $p$-Wert. Wir erkennen, dass die Mittelwertsdifferenz bei $d$= -0.065 liegt, mit zugehörigen $t$-Wert t(1998)=-1.4456 und $p$-Wert p=0.1484, somit ist dieser Mittelwertsvergleich auf dem 5% Niveau nicht signifikant ($p$>.05).
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -0.15317201 0.02317988
## sample estimates:
## mean of x mean of y
## -0.002997332 0.061998736
zeigt uns die Alternativhypothese ($H_1:d\neq0$), das Konfidenzintervall der Mittelwertsdifferenz sowie die Mittelwerte in den beiden Gruppen ($\bar{X}=$ -0.0029973, $\bar{Y}=$ 0.0619987). Somit ist auch die Null-Hypothese klar: $H_0:d=0$.
Wir können den Test auch als R Objekt abspeichern und ähnlich wie den Datensätzen anschließend diese Informationen entlocken. Dazu müssen wir das ausgegebene Objekt t.test lediglich einem Namen zuordnen und anschließend mit $ ansprechen. Wieso nennen wir das Objekt nicht einfach ttest. Wenn wir names darauf anwenden, sehen wir alle Namen, die wir hinter $ schreiben können:
ttest <- t.test(X, Y, var.equal = T)
names(ttest) # alle möglichen Argumente, die wir diesem Objekt entlocken können
## [1] "statistic" "parameter" "p.value" "conf.int" "estimate" "null.value"
## [7] "stderr" "alternative" "method" "data.name"
ttest$statistic # (empirischer) t-Wert
## t
## -1.4456
ttest$p.value # zugehöriger p-Wert
## [1] 0.1484462
Da die Null-Hypothese nicht verworfen wird, bedeutet dies, dass wir weiterhin annehmen, dass die Mittelwertsdifferenz in der Population tatsächlich 0 ist. Die Mittelwertsdifferenz in der Stichprobe ist offensichtlich nicht 0. Allerdings liegt sie so nah an der 0, dass diese Abweichung wahrscheinlich durch Zufall passiert ist und sich nicht auf die Population verallgemeinern lässt (siehe als Wiederholung Eid, et al., 2017, Kapitel 11.1 für den $t$-Test und Kapitel 8 für Inferenzstatistikgrundlagen und Hypothesentests, bzw. Agresti, & Finlay, 2013, Kapitel 6).
Verteilung unter $H_0$
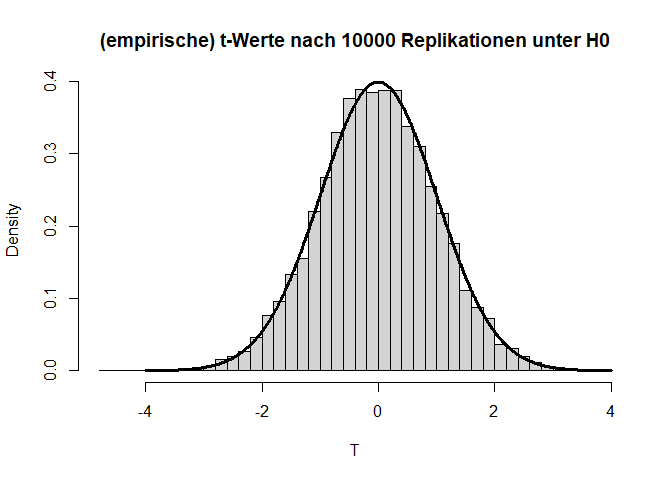
Wenn wir dieses Experiment nun ganz häufig wiederholen, dann sollte die Teststatistik $T=\frac{\bar{X}-\bar{Y}}{\sigma_p}$ (wobei $\bar{X}$ und $\bar{Y}$ die Mittelwerte von $X$ und $Y$ sind und $\sigma_p$ die gepoolte Standardabweichung beschreibt) der $t$-Verteilung folgen, welcher wir dann den $p$-Wert ablesen können. Das ist nämlich genau die Idee hinter dem inferenzstatistischen Testen. Hierbei entspricht die Teststatistik dem empirischen $t$-Wert. Die Bezeichnung kann also austauschbar verwendet werden! Wenn Sie sich für den dahinterliegenden Code interessieren, so können Sie diesen im Appendix A nachlesen.
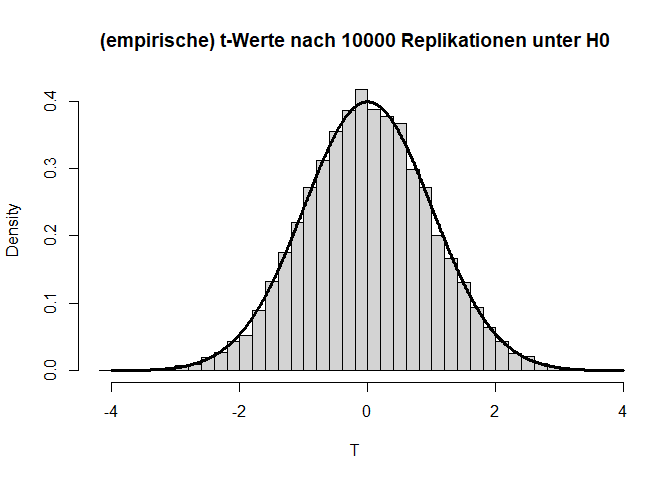
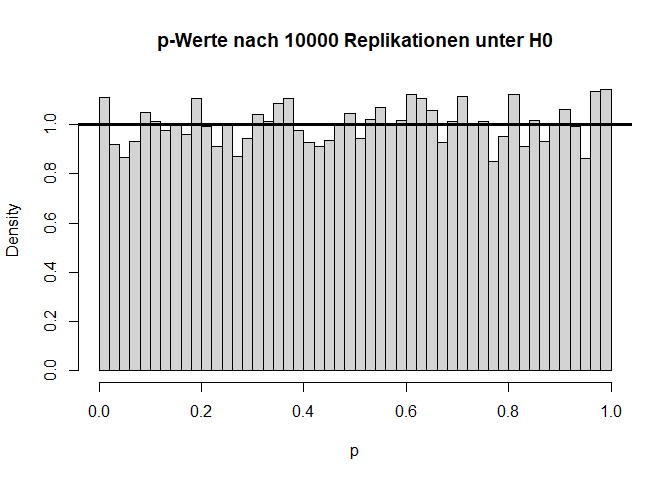
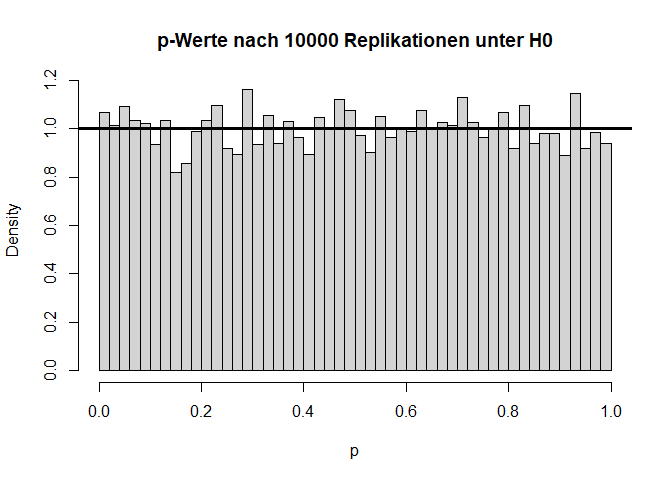
Die beiden Histogramme zeigen die empirische Verteilung der $t$- und $p$-Werte unter der $H_0$-Hypothese nach 10000 (unabhängigen) Wiederholungen und die angenommene Verteilung (fette durchgezogene schwarze Linie). Von den $p$-Werten wird erwartet, dass sie sich gleich (uniform) auf das Intervall zwischen 0 und 1 verteilen. Somit landen dann nur 5% der $p$-Werte mit zugehörig großen Teststatistiken (zufällig) im Bereich $p<0.05$.
Verteilung unter $H_1$
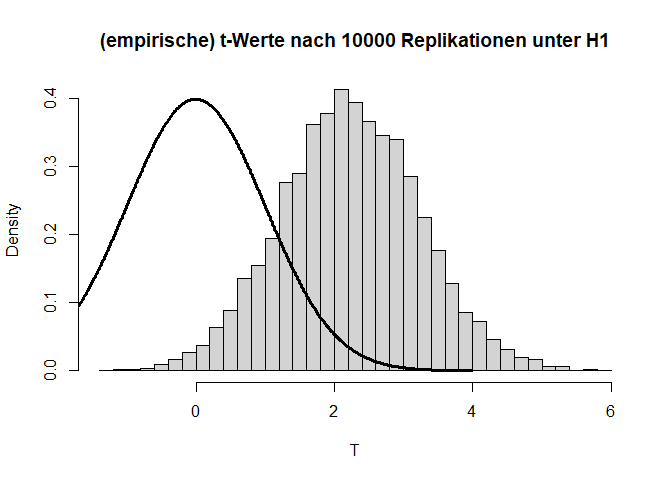
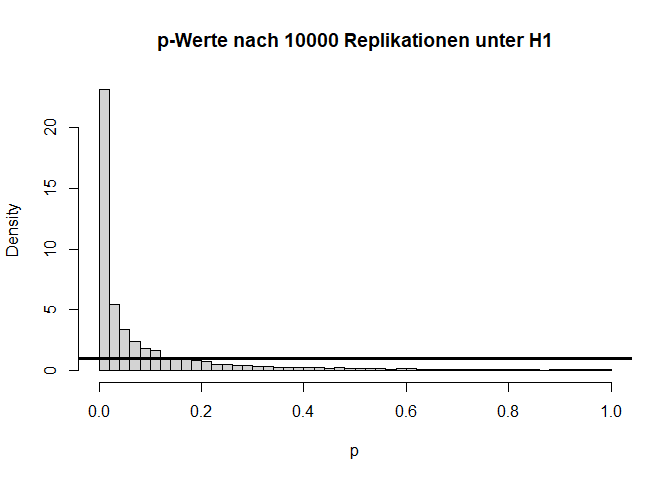
Angenommen die $H_0$-Hypothese gilt nicht und es liegt tatsächlich eine Mittelwertsdifferenz von bspw. $d=0.1$ ($H_1: \mu_1 - \mu_2 = 0.1$) vor, dann hat dies folgende Auswirkungen auf die Verteilung der Teststatistik und die zugehörigen $p$-Werte:
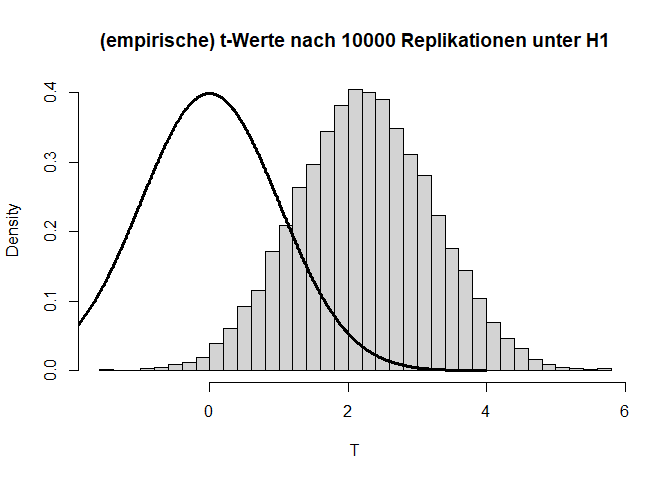
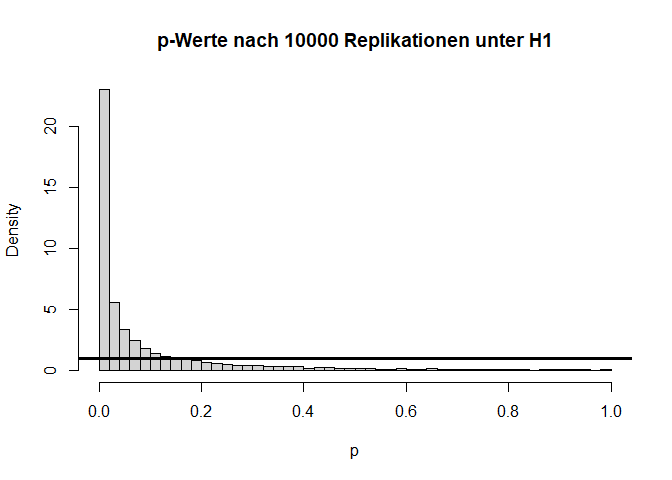
Wir sehen sehr deutlich, dass die Teststatistik $T$ deutlich nach rechts verschoben ist und nicht mehr zur theoretischen Verteilung unter der $H_0$-Hypothese passt. Auch die $p$-Werte sind alles andere als gleichverteilt. Folglich sprechen extreme $t$-Werte gegen die Null-Hypothese, weswegen wir diese verwerfen, wenn wir einen extremen Wert beobachten. Hier liegen 60.99% der $p$-Werte unterhalb von $0.05$. Dies wird auch als Power (siehe im Kapitel 8 in Eid, et al., 2017 Wiederholungen der Begriffe Power und $\alpha$-Fehler) bezeichnet. Somit hat der $t$-Test für eine Mittelwertsdifferenz von $d$=.1 und eine Stichprobengröße von insgesamt n = 2000 eine Power von rund 60.99%. Dies bedeutet, dass in diesem Fall die $H_0$ in 60.99% der Fälle richtigerweise verworfen wird. Schauen wir uns die Power der $t$-Tests einmal für verschiedene Stichprobengröße ($n$) und Mittelwertsdifferenzen ($d$) an:
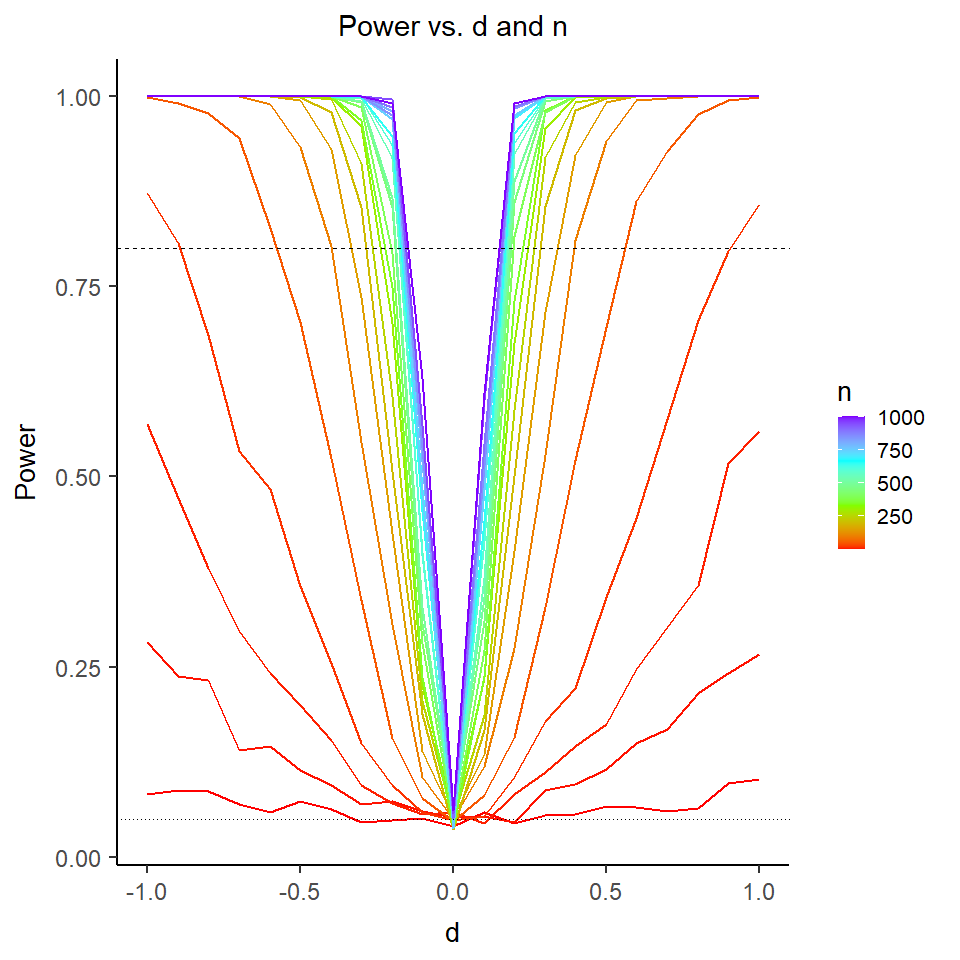
Auf der x-Achse sehen wir unterschiedliche Mittelwertsdifferenzen. Auf der y-Achse ist die Power abgetragen. Die Farbe kodiert die Stichprobengröße. Die horizontal gepunktete Linie zeigt eine Power von 5% (also das vorgegebene $\alpha$-Niveau) an und die horizontal gestrichelte Linie zeigt eine Power von 80% an.
Wir sehen sehr deutlich, dass für alle Stichprobengrößen (farblich kodiert) von $n=2$ bis $n=1000$ (pro Gruppe) die Power bei einer Mittelwertsdifferenz von 0 gerade bei ca. 5% liegt. Die Mittelwertsdifferenz von 0 beschreibt gerade die Null-Hypothese, dass es keinen Mittelwertsunterschied gibt. In diesem Fall beobachten wir den $\alpha$-Fehler dieses Tests, nämlich die Wahrscheinlichkeit, zufällig die Null-Hypothese zu verwerfen, obwohl diese gilt. Die Power von 5% war somit zu erwarten, da dies gerade das $\alpha$-Niveau dieses Tests ist (welches wir uns auch vorher so vorgegeben haben). In der Regel wird $\alpha = .05$ gewählt.
Was wir auch erkennen ist, dass für sehr große Stichproben die Power dieses Tests schon für sehr kleine Mittelwertsdifferenzen groß ist (idealerweise sprechen Methodiker von einem Test mit hinreichender Power, wenn diese größer als 80% ist). Dieses Beispiel zeigt sehr schön auf, dass damit die Power (also die Wahrscheinlichkeit einen Effekt zu finden, wenn dieser da ist) hoch ist, es einen Effekt geben muss (was sehr trivial klingt und es eigentlich auch ist) und zudem die Stichprobengröße hinreichend groß sein muss. Mit hinreichend groß hält sich der/die Statistiker/in die Hintertür offen zu sagen, dass: a) im Falle eines kleinen Effekts die Stichprobengröße eben sehr groß sein muss, um diesen zu erkennen und b) bei Vorliegen eines sehr großen Effekts schon eine kleine Stichprobengröße ausreicht, um den Effekt mit hinreichender Wahrscheinlichkeit auch als solchen zu identifizieren. Das ist auch der Grund, warum es Poweranalysen gibt, mit welchen bestimmt werden kann, wie groß die Stichprobengröße sein muss, um bei einem vorgegebenem erwarteten Effekt hinreichende Power zu haben, diese auch zu entdecken. Für sehr einfache Tests gibt es auch geschlossene Formeln, um bspw. Stichprobengröße zu finden, mit welcher vorgegebene Effekte mit hinreichend großer Wahrscheinlichkeit aufgedeckt werden können. Eines dieser Pakete ist das pwr Paket. Mit diesem können wir die oben dargestellte Grafik auch ohne Simulation (nur mit Formeln) erstellen (um das Ganze übersichtlicher zu gestalten, sind hier mehr Stichprobengrößen abgetragen, allerdings werden nur Stichproben von 2 bis 500 im Plot integriert):
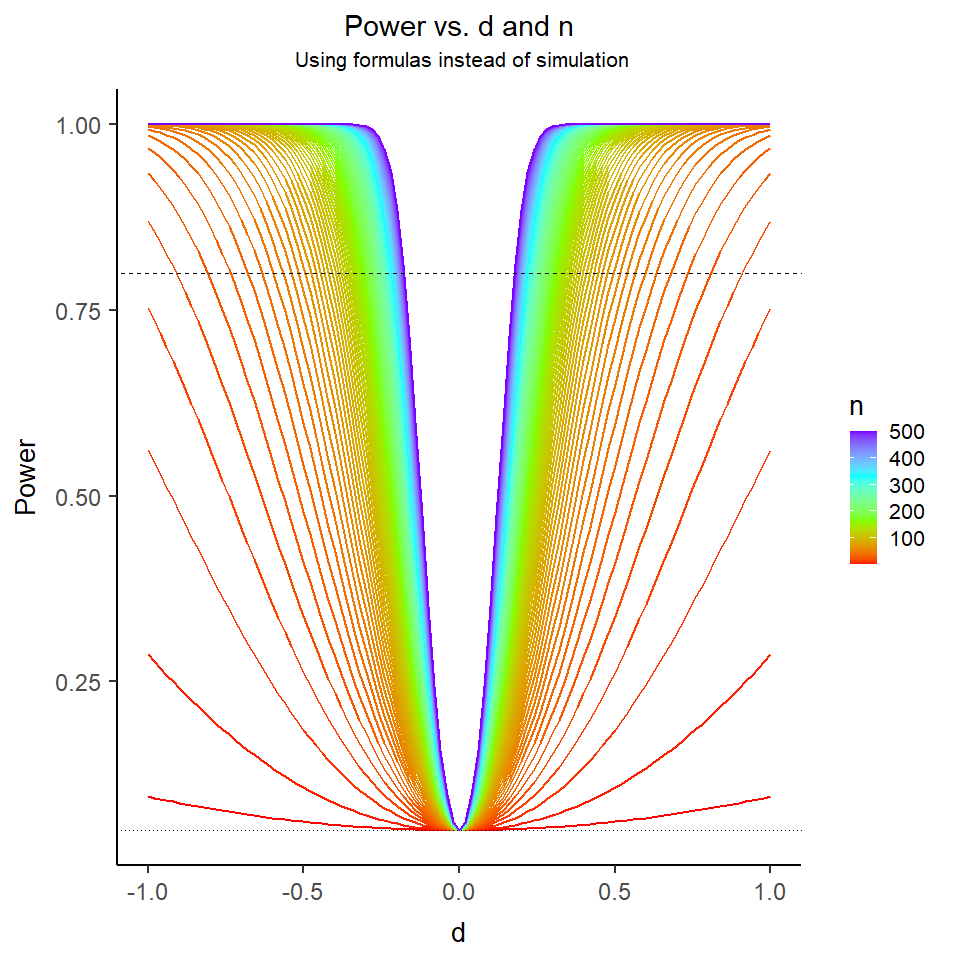
Wir erkennen deutlich, wie “smooth” der Plot eigentlich aussehen sollte. Daran ist ersichtlich, dass unsere Simulation wesentlich mehr Wiederholung benötigt, um ein solches Bild abzugeben.
Für Interessierte ist in Appendix B ein kleiner Exkurs in pwr dargestellt.
Verstöße gegen die Modellannahmen
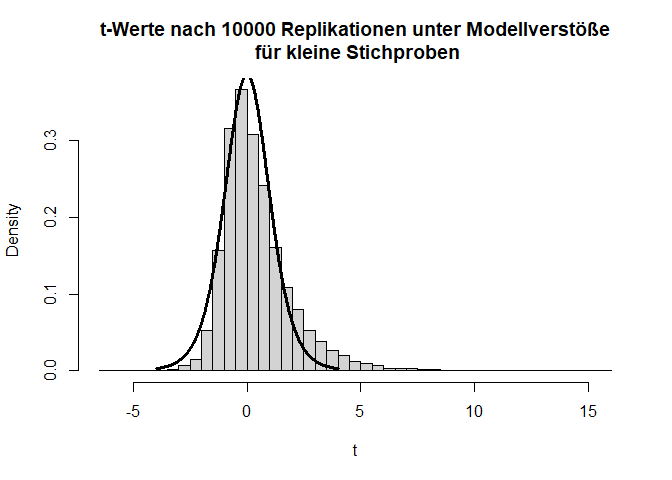
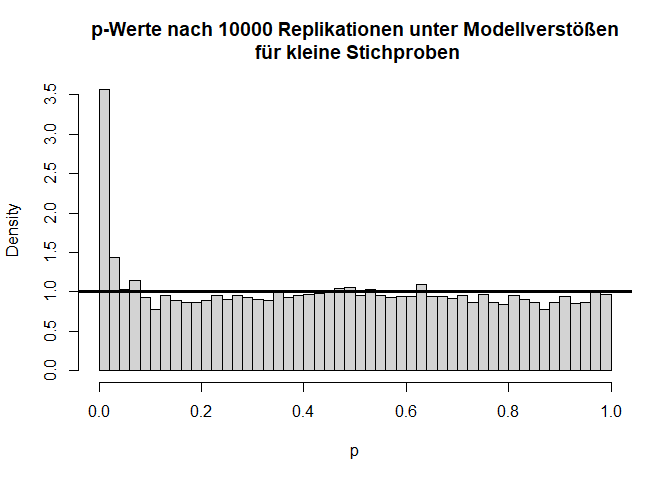
Liegen andere Verstöße gegen die Modellannahmen vor, dann kann es fälschlicherweise zu signifikanten Ergebnissen kommen, obwohl es in der Population gar keinen Effekt gibt. Dies ist oft bei kleinen Stichproben ein Problem. Nehmen wir beispielsweise an, dass die beiden Gruppen sehr gegenläufig schief verteilt sind.
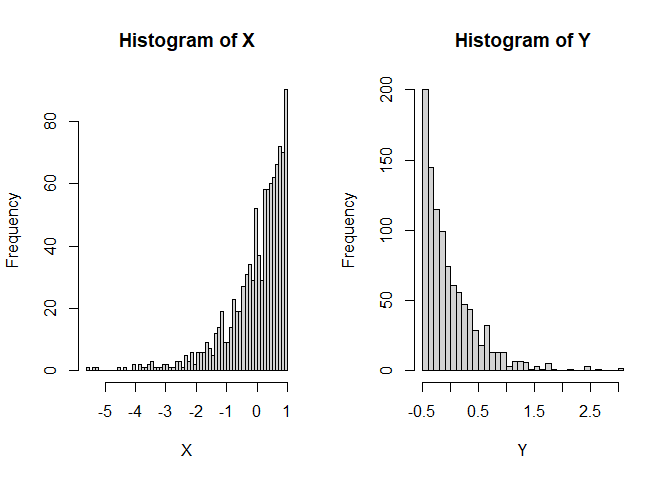
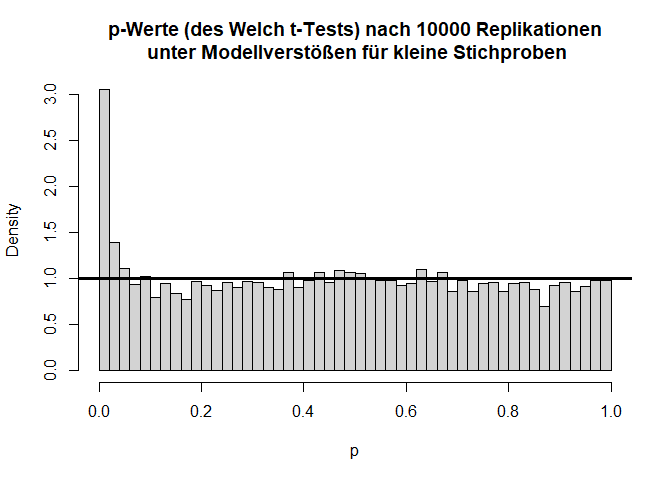
Hier gilt zwar die Null-Hypothese, da beide Verteilungen einen theoretischen Mittelwert von 0 haben, aber die Varianzen unterscheiden sich (was im Histogramm durch die extremeren Werte entlang der x-Achse zu erkennen ist) und die Variablen sind offensichtlich nicht normalverteilt. Schauen wir uns nun die Power des $t$-Tests für eine sehr kleine Stichprobe von 5 pro Gruppe an:
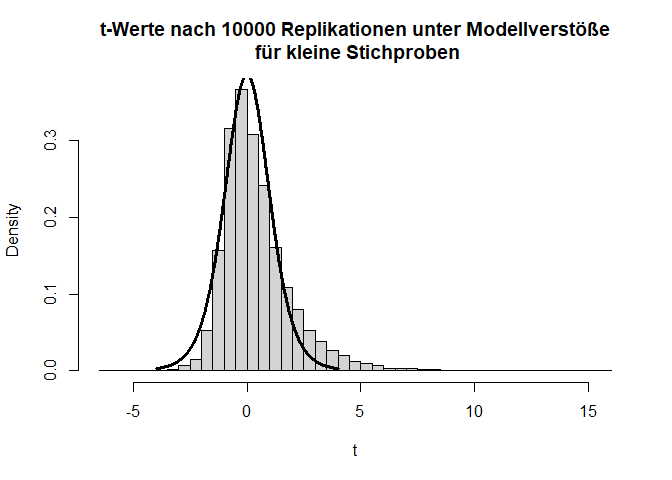
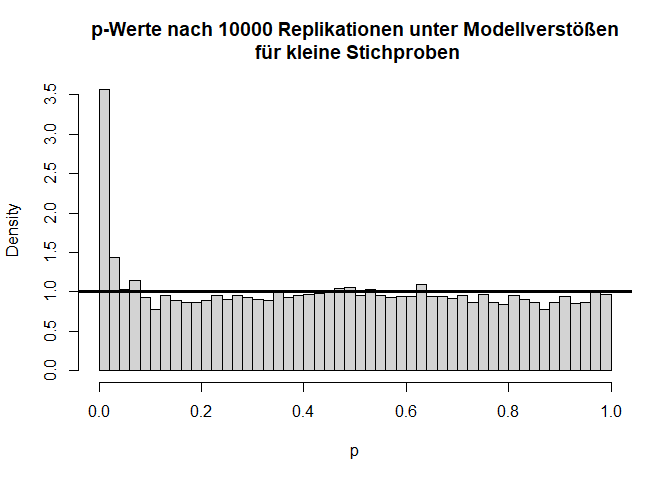
Insgesamt sieht die Verteilung der $T$-Werte einigermaßen in Ordnung, wenn auch etwas schief, aus. Doch bei den $p$-Werten fällt auf, dass die Null-Hypothese zu häufig verworfen wird, nämlich insgesamt in 11.11% der Fälle (durch Zufall, da die Null-Hypothese eigentlich gilt!); also doppelt so häufig wie von uns vorgegeben! Glücklicherweise ist der $t$-Test relativ robust, was daran zu erkennen ist, dass, wenn die Stichprobengröße für dieses Beispiel bei 50 oder gar höher liegt (pro Gruppe), das $\alpha$-Niveau schon wieder einigermaßen eingehalten wird. Außerdem gibt es geeignetere Tests zum Untersuchen von Mittelwertsunterschieden zweier Stichproben als den $t$-Test - nämlich den Welch-Test. Dies ist eine Erweiterung des $t$-Tests für ungleiche Varianzen. Dieser ist auch der Default in R. Wir rechnen ihn, indem wir nicht länger var.equal = T in t.test spezifizieren. Der Output ändert sich bis auf die Namensänderung kaum - die Freiheitsgrade des Tests werden korrigiert, um auf die ungleichen Varianzen zu reagieren (dies bedeutet, immer wenn die Freiheitsgrade nicht einfach $n-2$ sind, dann wurde der Welch-Test gerechnet; insbesondere Kommazahlen als $df$ sind möglich). Jedoch bringt diese Erweiterung ebenfalls nur für größere Stichproben etwas. Die analoge Simulationsstudie können Sie Appendix A entnehmen.
Diese Sitzung sollte als Einführung in R, Vorbereitung für die Regressionssitzung und kleine Wiederholung einiger wichtiger inferenzstatistischer Begriffe (wie etwa $\alpha$-Fehler, Power, Hypothese etc.) fungieren. Wie wir Histogramme erstellen und Verteilungen vergleichen, erfahren wir in der nächsten Sitzung im Rahmen der Voraussetzungen der Regressionsanalyse und Ausreißerdiagnostik.
Eine Übersicht über Matrixalgebra in R erhalten Sie in Appendix C.
Eine schöne Einleitung in Plots in R, wie man diese erstellt, bearbeitet und abspeichert, können Sie in den Unterlagen aus dem Bachelor (PsyBsc2) zu R Deskriptivstatistik nachlesen, wenn Sie denn wollen.
Appendix
Appendix A
Analoge Simulationsstudie
Hier ist der Code für einige Grafiken und Simulationen dargestellt. Dies geht natürlich über den Stoff des Seminars hinaus und ist nur für Interessierte bestimmt.
Verteilung unter $H_0$
ts <- c(); ps <- c() # wir brauchen zunächst Vektoren, in die wir die t-Werte und die p-Werte hineinschreiben können
for(i in 1:10000)
{
X <- rnorm(n = 1000, mean = 0, sd = 1)
Y <- rnorm(n = 1000, mean = 0, sd = 1)
ttest <- t.test(X, Y, var.equal = T)
ts <- c(ts, ttest$statistic) # nehme den Vektor ts und verlängere ihn um den neuen t-Wert
ps <- c(ps, ttest$p.value) # nehme den Vektor ps und verlängere ihn um den neuen p-Wert
}
hist(ts, main = "(empirische) t-Werte nach 10000 Replikationen unter H0",
xlab = "T", freq = F, breaks = 50)
lines(x = seq(-4,4,0.01), dt(x = seq(-4,4,0.01), df = ttest$parameter),
lwd = 3)
hist(ps, main = "p-Werte nach 10000 Replikationen unter H0",
xlab = "p", freq = F, breaks = 50)
abline(a = 1, b = 0, lwd = 3)
Verteilung unter $H_1$
ts <- c(); ps <- c() # wir brauchen zunächst Vektoren, in die wir die t-Werte und die p-Werte hineinschreiben können
for(i in 1:10000)
{
X <- rnorm(n = 1000, mean = 0, sd = 1)
Y <- -0.1 + rnorm(n = 1000, mean = 0, sd = 1) # Mittelwertsdifferenz ist 0.1
ttest <- t.test(X, Y, var.equal = T)
ts <- c(ts, ttest$statistic) # nehme den Vektor ts und verlängere ihn um den neuen t-Wert
ps <- c(ps, ttest$p.value) # nehme den Vektor ps und verlängere ihn um den neuen p-Wert
}
hist(ts, main = "(empirische) t-Werte nach 10000 Replikationen unter H1",
xlab = "T", freq = F, breaks = 50)
lines(x = seq(-4,4,0.01), dt(x = seq(-4,4,0.01), df = ttest$parameter),
lwd = 3)
hist(ps, main = "p-Werte nach 10000 Replikationen unter H1",
xlab = "p", freq = F, breaks = 50)
abline(a = 1, b = 0, lwd = 3)
Verteilung unter $H_0$ mit Modellverstößen
set.seed(1)
ts <- c(); ps <- c() # wir brauchen zunächst Vektoren, in die wir die t-Werte und die p-Werte hineinschreiben können
for(i in 1:10000)
{
X <- -rexp(n = 5, rate = 1) # simuliere Exponentialverteilung zur Rate 1 mit n = 5
X <- X + 1 # zentriere, sodass der Populationsmittelwert wieder 0 ist
Y <- rexp(n = 5, rate = 2) # simuliere Exponentialverteilung zur Rate 2 mit n = 5
Y <- Y - 1/2 # zentriere, sodass der Populationsmittelwert wieder 0 ist
ttest <- t.test(X, Y, var.equal = T)
ts <- c(ts, ttest$statistic) # nehme den Vektor ts und verlängere ihn um den neuen t-Wert
ps <- c(ps, ttest$p.value) # nehme den Vektor ps und verlängere ihn um den neuen p-Wert
}
hist(ts, main = "t-Werte nach 10000 Replikationen unter Modellverstöße\n für kleine Stichproben",
xlab = "t", freq = F, breaks = 50)
lines(x = seq(-4,4,0.01), dt(x = seq(-4,4,0.01), df = ttest$parameter),
lwd = 3)
hist(ps, main = "p-Werte nach 10000 Replikationen unter Modellverstößen\n für kleine Stichproben",
xlab = "p", freq = F, breaks = 50)
abline(a = 1, b = 0, lwd = 3)
Verteilung unter $H_0$ mit Modellverstößen: Welch-Test
set.seed(1)
ts <- c(); ps <- c() # wir brauchen zunächst Vektoren, in die wir die t-Werte und die p-Werte hineinschreiben können
for(i in 1:10000)
{
X <- -rexp(n = 5, rate = 1) # simuliere Exponentialverteilung zur Rate 1 mit n = 5
X <- X + 1 # zentriere, sodass der Populationsmittelwert wieder 0 ist
Y <- rexp(n = 5, rate = 2) # simuliere Exponentialverteilung zur Rate 2 mit n = 5
Y <- Y - 1/2 # zentriere, sodass der Populationsmittelwert wieder 0 ist
ttest <- t.test(X, Y) # Welch Test
ts <- c(ts, ttest$statistic) # nehme den Vektor ts und verlängere ihn um den neuen t-Wert
ps <- c(ps, ttest$p.value) # nehme den Vektor ps und verlängere ihn um den neuen p-Wert
}
hist(ts, main = "t-Werte (des Welch t-Tests) nach 10000 Replikationen\n unter Modellverstöße für kleine Stichproben",
xlab = "t", freq = F, breaks = 50)
lines(x = seq(-4,4,0.01), dt(x = seq(-4,4,0.01), df = ttest$parameter),
lwd = 3)
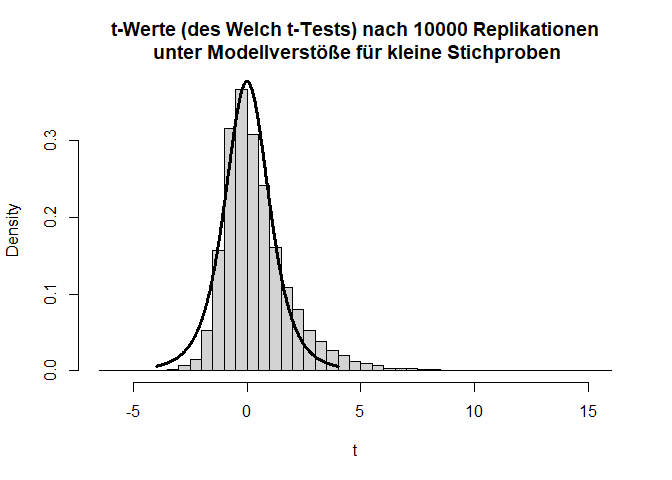
hist(ps, main = "p-Werte (des Welch t-Tests) nach 10000 Replikationen\n unter Modellverstößen für kleine Stichproben",
xlab = "p", freq = F, breaks = 50)
abline(a = 1, b = 0, lwd = 3)
Verteilung unter $H_0$ mit Modellverstößen mit größerer Stichprobe: Welch-Test
set.seed(1234)
ts <- c(); ps <- c() # wir brauchen zunächst Vektoren, in die wir die t-Werte und die p-Werte hineinschreiben können
for(i in 1:10000)
{
X <- -rexp(n = 100, rate = 1) # simuliere Exponentialverteilung zur Rate 1 mit n = 100
X <- X + 1 # zentriere, sodass der Populationsmittelwert wieder 0 ist
Y <- rexp(n = 100, rate = 2) # simuliere Exponentialverteilung zur Rate 2 mit n = 100
Y <- Y - 1/2 # zentriere, sodass der Populationsmittelwert wieder 0 ist
ttest <- t.test(X, Y) # Welch Test
ts <- c(ts, ttest$statistic) # nehme den Vektor ts und verlängere ihn um den neuen t-Wert
ps <- c(ps, ttest$p.value) # nehme den Vektor ps und verlängere ihn um den neuen p-Wert
}
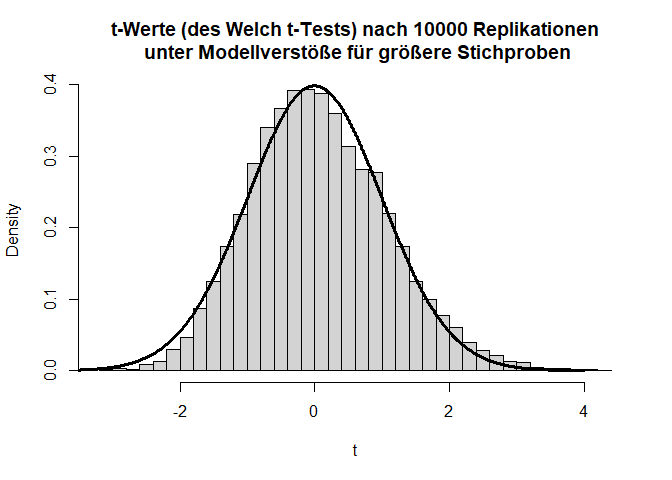
hist(ts, main = "t-Werte (des Welch t-Tests) nach 10000 Replikationen\n unter Modellverstöße für größere Stichproben",
xlab = "t", freq = F, breaks = 50)
lines(x = seq(-4,4,0.01), dt(x = seq(-4,4,0.01), df = ttest$parameter),
lwd = 3)
hist(ps, main = "p-Werte (des Welch t-Tests) nach 10000 Replikationen\n unter Modellverstößen für größere Stichproben",
xlab = "p", freq = F, breaks = 50)
abline(a = 1, b = 0, lwd = 3)
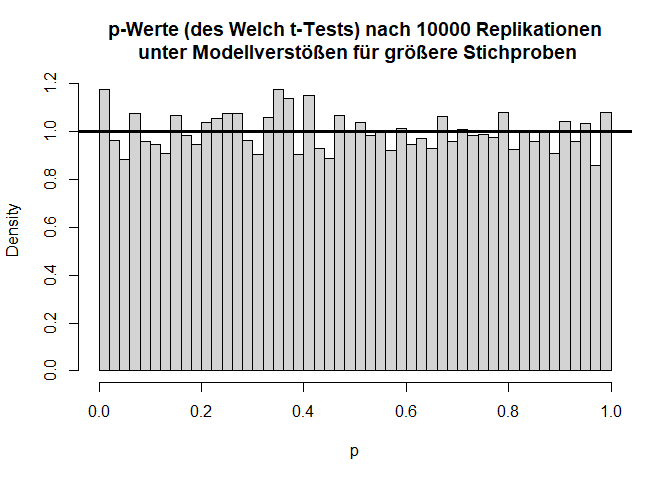
Für jeweils 100 Erhebungen pro Gruppe ist der Verstoß gegen die Normalverteilungsannahme bei ungleichen Varianzen für den Welch-Test fast zu vernachlässigen.
Appendix B
Poweranalysen: geschlossene Formeln
Für den $t$-Test lässt sich die Power auch über Formeln finden. Diese sind im pwr-Paket implementiert. Die Funktion pwr.t.test ist die Richtige. Sie nimmt zwei wichtige Argumente entgegen: n und d. Hierbei ist $n$ die Stichprobengröße und $d$ ist die Effektstärke nach Cohen:
$$d:=\frac{|\mu_1-\mu_2|}{\sigma},$$ wobei $\mu_1$ und $\mu_2$ die beiden Mittelwerte in den Gruppen sind und $\sigma$ ist die wahre Standardabweichung über die beiden Gruppen hinweg. Dadurch, dass bei uns die Varianz jeweils 1 in den Gruppen war, hatten wir durch Zufall auch oben die Effektstärke nach Cohen gewählt! Die Grafiken sind also durchaus vergleichbar. Wir schauen uns die Power von einer Effektstärke von 0.1 bei einer Stichprobengröße von 1000 an:
library(pwr)
pwr.t.test(n = 1000, d = 0.1)
##
## Two-sample t test power calculation
##
## n = 1000
## d = 0.1
## sig.level = 0.05
## power = 0.6083667
## alternative = two.sided
##
## NOTE: n is number in *each* group
Die Power liegt bei 0.6083, also bei 60.83%. Sie ist damit also gar nicht so verschieden zu unseren 60.99%. Das liegt hier nun daran, dass die Stichproben sehr groß sind und damit die Asymptotik gut greift. Bei kleinen Stichproben sind sehr viele Replikationen nötig, damit die Power auch dort nahe der theoretischen Power liegt! Mehr Informationen zum pwr-Paket (bspw. Power von ANOVAs) finden sie hier.
Appendix C
Matrixalgebra
Matrixalgebra wird bspw. auch in Eid, et al. (2017) im Anhang B: Matrixalgebra ab Seite 1051 behandelt.
R ist eine vektorbasierte Programmiersprache, was bedeutet, dass möglichst viel mit Vektor- oder Matrixoperationen durchgeführt werden soll. Um davon Gebrauch zu machen, müssen wir uns mit diesen Operationen vertraut machen:
Vektoren
Dazu seien X und Y zwei Vektoren, die wir mit dem Zuordnungspfeil <- erstellen und mit der Vektorfunktion c() erstellen:
X <- c(1, 2, 3)
Y <- c(10, 8, 6)
Wir können auf Elemente eines Vektor mit eckigen Klammern zugreifen. Bspw. erhalten wir das 2. Element von Y (also quasi $y_2$) mit
Y[2]
## [1] 8
Elementeweise Additionen funktionieren super simpel, indem wir sie einfach mit + verknüpfen. Auch Subtraktionen oder skalare Multiplikationen funktionieren so:
X + Y # Addition
## [1] 11 10 9
X - Y # Subtraktion
## [1] -9 -6 -3
3*X # Skalare Multiplikation
## [1] 3 6 9
1/2*X
## [1] 0.5 1.0 1.5
Wenn X und Y nicht die selbe Länge haben, ist es in R oft so, dass die Vektoren künstlich verlängert werden, um verrechnet zu werden. Dies sollten wir immer im Hinterkopf behalten.
Z <- c(1:6) # Zahlen 1 bis 6
Z + Y
## [1] 11 10 9 14 13 12
Z ist hier doppelt so lang wie Y, sodass in der Addition Y einfach zweimal hintereinander geschrieben wird, damit die Addition möglich ist, denn eine Addition bei Vektoren (und auch Matrizen) funktioniert nur, wenn die beiden Elemente das gleiche Format haben!
Wenn wir zwei Vektoren miteinander multiplizieren, so entsteht kein Matrixprodukt, sondern eine elementeweise Multiplikation:
X*Y # elementeweise Multiplikation
## [1] 10 16 18
Matrizen
Die gerade behandelten Vektoren können wir ganz leicht zu einer Matrix machen, indem wir den Befehl as.matrix bspw. auf X anwenden. Dieser Befehl erzeugt eine 3x1 Matrix - also eigentlich einen Spaltenvektor.
as.matrix(X)
## [,1]
## [1,] 1
## [2,] 2
## [3,] 3
Wir können die beiden Vektoren auch zu einer Matrix kombinieren, indem wir sie bspw. als zwei Zeilenvektoren mit dem Befehl cbind (was für column binding steht) zusammenfügen - genauso geht dies auch mit rbind (was für row binding steht):
A <- cbind(X, Y)
A
## X Y
## [1,] 1 10
## [2,] 2 8
## [3,] 3 6
B <- rbind(X, Y)
B
## [,1] [,2] [,3]
## X 1 2 3
## Y 10 8 6
Wir können nun bspw. den Eintrag $A_{12}$ herauslesen via [1, 2], wobei der erste Eintrag immer für die Zeile und der 2. für die Spalte steht:
A[1, 2] # Eintrag 1. Zeile 2. Spalte in A
## Y
## 10
Eine ganze Zeile oder Spalte erhalten wir, indem wir eines der Elemente in der Indizierung frei lassen:
A[1, ] # 1. Zeile
## X Y
## 1 10
A[, 2] # 2. Spalte
## [1] 10 8 6
Wir können eine Matrix transponieren; also Zeilen und Spalten vertauschen; indem wir den Befehl t() auf die Matrix anwenden:
A
## X Y
## [1,] 1 10
## [2,] 2 8
## [3,] 3 6
t(A)
## [,1] [,2] [,3]
## X 1 2 3
## Y 10 8 6
B
## [,1] [,2] [,3]
## X 1 2 3
## Y 10 8 6
Wir erkennen, dass die Matrix B gerade die Transponierte von A ist! Die beiden Matrizen A und B lassen sich nicht addieren, da sie nicht das richtige Format haben, während skalare Multiplikation immer funktioniert:
A + B
## Error in A + B : non-conformable arrays
A * 2
## X Y
## [1,] 2 20
## [2,] 4 16
## [3,] 6 12
Da die beiden Matrizen gerade das Format 3x2 und 2x3 haben, lassen sie sich aber als Matrixprodukt verrechnen. Der Operator hierfür heißt %*% (verwenden wir stattdessen *, so wird eine elementenweise Multiplikation durchgeführt, was etwas komplett anderes ist!):
A %*% B # Matrixprodukt A*B
## [,1] [,2] [,3]
## [1,] 101 82 63
## [2,] 82 68 54
## [3,] 63 54 45
B %*% A # Matrixprodukt B*A
## X Y
## X 14 44
## Y 44 200
An den Ergebnissen erkennen wir auch, dass Matrixprodukte nicht kommutativ sind, also die Reihenfolge wichtig ist in der (matrix-)multipliziert wird.
Spezielle Matrizen
Eine quadratische Matrix hat genauso viele Zeilen wie Spalten. Eine wichtige quadratische Matrix ist die Einheitsmatrix $I$, welche nur 1en auf der Diagonalen und sonst 0en hat. Diese ist gerade das Elemente, mit welchem wir getrost multiplizieren können (falls die Dimensionen stimmen), weil dann nichts passiert (wie Multiplikation mit 1 bei Zahlen). Wir erhalten sie mit diag, was eigentlich eine (quadratische) Diagonalmatrix erzeugt mit beliebigen Elementen auf der Diagonalen:
diag(3) # Einheitsmatrix 3x3
## [,1] [,2] [,3]
## [1,] 1 0 0
## [2,] 0 1 0
## [3,] 0 0 1
diag(1:3) # Diagonalmatrix mit Elementen 1,2,3 auf der Diagonalen
## [,1] [,2] [,3]
## [1,] 1 0 0
## [2,] 0 2 0
## [3,] 0 0 3
Wir können eine Matrix mit dem matrix Befehl auch mit Hand füllen. Diesem übergeben wir einen Vektor und die Dimensionen der Matrix (data werden die Daten, die wir in die Matrix schreiben wollen übergeben, nrow und ncol bestimmen die Anzahl der Zeilen und Spalten und mit byrow = T zeigen wir an, dass wir die Matrix zeilenweise gefüllt bekommen möchten):
C <- matrix(data = c(1:9), nrow = 3, ncol = 3, byrow = T)
C
## [,1] [,2] [,3]
## [1,] 1 2 3
## [2,] 4 5 6
## [3,] 7 8 9
Wir können mit diag auch wieder die Diagonalelemente einer Matrix erfahren:
diag(C)
## [1] 1 5 9
Determinanten und Invertierung
Die Inverse, also jenes Element, mit welchem wir (matrix-)multiplizieren müssen, um die Einheitsmatrix zu erhalten, lässt sich in R mit dem solve Befehl erhalten (dies geht nur bei quadratischen Matrizen):
solve(C)
## Error in solve.default(C) :
## system is computationally singular: reciprocal condition number = 2.59052e-18
Die Matrix C lässt sich nicht invertieren, da sie singulär ist und damit nicht invertierbar. Dies bedeutet, dass es lineare Abhängigkeiten der Zeilen bzw. Spalten gibt. Wir können dies explizit prüfen, indem wir die Determinante bestimmen mit det:
det(C)
## [1] 6.661338e-16
round(det(C), 14)
## [1] 0
Mit round runden wir das Ergebnis auf die 14. Nachkommastelle. Eine Matrix ist genau dann invertierbar (also regulär im Vergleich zu singulär), wenn die Determinante dieser (quadratischen) Matrix nicht Null ist. Lineare Abhängigkeit bedeutet, dass die Zeilen oder Spalten durch Addition, Subtraktion und skalare Multiplikationen auseinander hervorgehen. Wir erkennen die lineare Abhängigkeit zwischen den Spalten, indem wir von der 2. Spalte die 1. Spalte abziehen und anschließend dies auf die 2. Spalte addieren - also de facto $2*2.Spalte - 1. Spalte$ rechnen:
2*C[, 2] - C[, 1] # 2*2.Spalte - 1. Spalte rechnen ist gleich
## [1] 3 6 9
C[, 3] # 3. Spalte
## [1] 3 6 9
Hätten wir C^-1 gerechnet, so hätten wir eine elementeweise Invertierung durchgeführt:
C^-1
## [,1] [,2] [,3]
## [1,] 1.0000000 0.500 0.3333333
## [2,] 0.2500000 0.200 0.1666667
## [3,] 0.1428571 0.125 0.1111111
C^-1 %*% C # ist nicht die Einheitsmatrix
## [,1] [,2] [,3]
## [1,] 5.333333 7.166667 9.000000
## [2,] 2.216667 2.833333 3.450000
## [3,] 1.420635 1.799603 2.178571
C^-1 * C # elementeweise ergibt überall 1 - ist immer noch nicht die Einheitsmatrix!
## [,1] [,2] [,3]
## [1,] 1 1 1
## [2,] 1 1 1
## [3,] 1 1 1
Dies bedeutet, dass C^-1 in R nicht die Invertierung betitelt sondern solve!
Betrachten wir nun eine invertierbare Matrix D:
D <- matrix(c(1, 0, 0,
1, 1, 1,
2, 4, 5), 3, 3, byrow = T)
det(D)
## [1] 1
Die Determinante von D ist 1. Somit können wir D invertieren. Das Produkt aus D mit seiner Inversen ergibt gerade die 3x3 Einheitsmatrix:
solve(D)
## [,1] [,2] [,3]
## [1,] 1 0 0
## [2,] -3 5 -1
## [3,] 2 -4 1
D %*% solve(D)
## [,1] [,2] [,3]
## [1,] 1 0 0
## [2,] 0 1 0
## [3,] 0 0 1
solve(D) %*% D
## [,1] [,2] [,3]
## [1,] 1 0 0
## [2,] 0 1 0
## [3,] 0 0 1
Das Produkt zwischen $D$ und $D^{-1}$ ist (ausnahmsweise) kommutativ: $DD^{-1}=D^{-1}D=I$.
Literatur
Agresti, A, & Finlay, B. (2013). Statistical methods for the social sciences. (Pearson new international edition, 4th edition). Harlow, Essex : Pearson Education Limited.
Eid, M., Gollwitzer, M., & Schmitt, M. (2017). Statistik und Forschungsmethoden (5. Auflage, 1. Auflage: 2010). Weinheim: Beltz.
- Blau hinterlegte Autor:innenangaben führen Sie direkt zur universitätsinternen Ressource.
](/media/header/chalkboard_equation.jpg)