In den letzten Sitzungen haben wir uns mit der Beantwortung von Zusammenhangsfragestellungen im Rahmen von Korrelation und Regression beschäftigt. Wie auch im letzten Semester werden wir uns nun mit Fragestellungen zum Unterschied beschäftigen. Dort haben wir bereits den t-Test kennen gelernt, mit dem Mittelwertsunterschiede zwischen zwei Gruppen untersucht werden können. Wenn wir nun mehr als zwei Gruppen miteinander vergleichen möchten, müssten wir mehrere t-Tests mit allen Kombinationen durchführen. Bei z. B. 3 Gruppen müssten wir $\binom{3}{2}$ - also in diesem Fall 3 t-Tests durchführen. Dies führt aber zu einer $\alpha$-Fehler-Inflation oder -Kumulierung.
Die Lösung für diese Problematik bietet die ANOVA, die uns die nächsten Wochen beschäftigen wird. Heute fokussieren wir uns zunächst auf ein Setting, in dem eine Gruppenvariable vorliegt, auf der sich die Personen untereinander unterscheiden (auch als between subject bezeichnet). Es handelt sich also hierbei zunächst um die Erweiterung des t-Tests für unabhängige Stichproben (Erweiterung des t-Tests für abhängige gibt es hier). Die Gruppierungsvariable kann beliebig viele Ausprägungen haben - wir bleiben im Beispiel bei 3 Stück. Wir nutzen heute also eine einfaktorielle ANOVA - die eine Gruppenvariable stellt den einen Faktor dar. Wie das Verfahren für mehrere Gruppenvariablen ist, wird in der nächsten Sitzung besprochen. Mehr zur einfaktoriellen ANOVA finden Sie in Eid, Gollwitzer und Schmitt (2017, Kapitel 13 und insb. 13.1 und folgend).
Die praktische Umsetzung in R soll anhand des Datensatzes conspiracy demonstriert werden, den wir also zunächst in unser Environment laden müssen. Dieser stammt aus einer Erhebung zur Validierung eines Fragebogens, der Skalenwerte enthält, die verschiedene Dimensionen von Verschwörungsglauben abbilden sollen.
Daten laden
Da die Rohdaten hier einige Informationen erhalten, die wir für die Durchführung des Tutorials nicht benötigen, stellen wir ein Aufbereitungsskript bereit, dass direkt eine Version des Datensatzes lädt, die für die Durchführung des Tutorials geeignet ist. Dieses kann mit dem source()-Befehl geladen und ausgeführt werden.
source("https://pandar.netlify.app/daten/Data_Processing_conspiracy.R")
Wie bereits erwähnt stammt der Datensatz aus einer Untersuchung zum Thema verschwörungstheoretische Überzeugungen. Beginnen wir mit einer kurzen Inspektion.
dim(conspiracy)
## [1] 2451 9
Der Datensatz enthält die Werte von 2451 Personen auf 9 Variablen.
head(conspiracy)
## edu urban gender age GM
## 1 not highschool suburban female 14 4.000000
## 2 college suburban female 26 2.000000
## 3 college rural male 25 5.000000
## 4 highschool suburban male 37 5.000000
## 5 college rural male 34 1.000000
## 6 highschool suburban male 17 3.333333
## GC EC PW CI
## 1 5.000000 4.666667 3.333333 4.666667
## 2 4.000000 1.000000 2.000000 3.333333
## 3 4.333333 1.000000 3.333333 4.666667
## 4 4.333333 2.333333 3.333333 4.666667
## 5 1.000000 1.000000 1.000000 1.000000
## 6 2.666667 3.000000 2.666667 3.666667
Die ersten vier Variablen enthalten Informationen über den demographischen Hintergrund der Personen: höchster Bildungsabschluss (edu), Typ des Wohnortes (urban), Geschlecht (gender) und Alter (age). Die fünf restlichen Variablen sind Skalenwerte bezüglich verschiedener Subdimensionen verschwörungstheoretischer Überzeugungen: GM (goverment malfeasance), GC (malevolent global conspiracies), EC (extraterrestrial cover-up), PW (personal well-being) und CI (control of information).
Einfaktorielle ANOVA
Die Durchführung der ANOVA benötigt natürlich Hypothesen, die uns bei der Auswertung interessieren. In unserem Beispiel soll untersucht werden, ob sich Personen je nach Ländlichkeit ihres Wohnortes (rural, suburban, urban - Variablenname urban) in der Überzeugung unterscheiden, inwiefern die Existenz von Außerirdischen (Skalenwert Variablenname EC) geheimgehalten wird (Beispielitem: Evidence of alien contact is being concealed from the public).
In der einfaktoriellen ANOVA wird die Gleichheit aller Gruppenmittelwerte als Nullhypothese postuliert. Dies bedeutet, dass sich Bewohner:innen des ländlichen Raums (rural), des vorstädtischen Raums (suburban) und der Stadt (urban) nicht hinsichtlich ihrer Zustimmung zur Verschwörungstheorie (EC) unterscheiden:
$H_0: \mu_{\text{rural}} = \mu_{\text{suburban}} = \mu_{\text{urban}}$
Im Rahmen der vorliegenden Daten müssen wir beachten, dass der Variablenname urban genauso gewählt wurde, wie eine seiner Ausprägungen (für den städtischen Raum). Für bestimmte Code-Abschnitte ist es wichtig, das im Hinterkopf zu behalten, um nicht verwirrt zu werden.
Bei der Alternativhypothese wird angenommen, dass sich mindestens zwei dieser Subgruppen im Mittel voneinander unterscheiden:
$H_1: \mu_j \neq \mu_k$ für mindestens ein Paar $(j, k)$ mit $j \neq k$
Wir benutzen hier die Indizes $j$ und $k$, um den Vergleich der Mittelwerte von zwei unterschiedlichen Subgruppen darzustellen. Für $j = 1$ und $k = 2$ könnte dies z. B. den Vergleich der Subgruppen “rural” und “suburban” anzeigen.
Bevor wir in die Testung einsteigen, müssen wir natürlich noch $\alpha$ festlegen - hier nehmen wir wie gewohnt $\alpha = .05$ an. Auch das Testen von Voraussetzungen ist natürlich wichtig - hierzu findet sich in Appendix A eine Übersicht.
Durchführung per Hand
Schauen wir uns zunächst an, wie wir die einfaktorielle ANOVA per Hand durchführen können. Dafür müssen wir uns zunächst die mathematischen Grundlagen klar machen. Der Kern der Berechnung basiert auf der Quadratsummenzerlegung. Hierbei werden Unterschiede/Variationen zwischen den Gruppen ($QS_{zw}$) und Unterschiede/Variationen innerhalb der Gruppen ($QS_{inn}$) getrennt betrachtet und bestimmt. Die Variation zwischen den Gruppen kann als bedingt durch die unterschiedliche Gruppenzugehörigkeit interpretiert werden, wobei die Variation innerhalb einer Gruppe von den Individuen, die der Gruppe zugehörig sind, bedingt wird. Diese beiden Bestandteile zusammen ergeben also die Gesamtvariation die dann als totale Quadratsumme ($QS_{tot}$) bezeichnet wird.
$$QS_{tot} = QS_{zw} + QS_{inn}$$
Die totale Quadratsumme als Repräsentant der Gesamtvariation kann auch als quadrierte Abstände aller Werte zum Gesamtmittelwert dargestellt werden. $i$ ist dabei der Index der Personen, $k$ der Index der Gruppe, $K$ die Gesamtanzahl der Gruppen und $n_k$ ist die Gruppengröße der k-ten Gruppen. $$QS_{tot} = \sum_{k = 1}^{K} \sum_{i = 1}^{n_k} (y_{ik}-\overline{y})^2$$
Auch wenn die Formel zunächst komplex aussieht, passiert hier nichts anderes, als dass vom Gesamtmittelwert der Wert jeder Person in der Stichprobe abgezogen wird. Die quadrierten Abweichungen werden dabei über alle Personen und Gruppen hinweg aufsummiert.
Die Quadratsumme zwischen den Gruppen repräsentiert die Abstände der Gruppenmittelwerte vom Gesamtmittelwert.
$$QS_{zw} = \sum_{k = 1}^{K} n_k* (\overline{y_k}-\overline{y})^2$$
Die Quadratsumme innerhalb der Gruppen ist die Summe der quadrierten Abweichungen aller Werte vom jeweiligen Gruppenmittelwert.
$$QS_{inn} = \sum_{k = 1}^{K} \sum_{i = 1}^{n_k} (y_{ik}-\overline{y_k})^2$$
Um die Quadratsummen der einfaktoriellen ANOVA per Hand zu bestimmen, müssen wir einige vorbereitende Schritte vollziehen. Zunächst brauchen wir die aggregate()-Funktion, die es erlaubt, eine zusammenfassende Statistik (wie Mittelwert oder Standardabweichung) für eine Variable getrennt nach verschiedenen Subgruppen zu berechnen. Dabei können wir in aggregate() die Schreibweise nutzen, die wir aus der Regression gewohnt sind - interessierende Variable ~ Gruppierungsvariable, data. Bei uns ist die interessierende Variable EC und die Gruppierungsvariable urban. Als zusätzliches Argument wird die Funktion übergeben, die in der Aggregation durchgeführt werden soll - also der Mittelwert durch mean().
# Gruppenmittelwerte ermitteln
y_mean_k <- aggregate(EC ~ urban, conspiracy, mean)
y_mean_k
## urban EC
## 1 rural 2.186667
## 2 suburban 2.140148
## 3 urban 2.296122
Unser erstelltes Objekt y_mean_k enthält nun die Mittelwerte der drei Gruppen. Die erste Spalte repräsentiert dabei die drei Ausprägungen der Variable urban - die zweite Spalte repräsentiert die empirischen Mittelwerte (und damit geschätzten Populationsmittelwerte) der Gruppe k auf der Variable EC. Der erste Spaltenname passt sehr gut - wir wollen aber hier die zweite Spalte noch neu benennen (EC_mean_k).
names(y_mean_k) <- c('urban', 'EC_mean_k')
Wir wollen nun die Mittelwerte der Gruppen zu unserem ursprünglichen Datensatz hinzufügen. Es soll eine neue Spalte entstehen, die jeder Person den Mittelwert der Gruppe zuweist, in der sie wohnhaft ist. Ein solches Zusammenführen ist mit der Funktion merge() möglich. Dafür müssen wir zunächst die beiden Datensätze angeben, die zusammengefügt werden sollen (conspiracy und y_mean_k). Natürlich müssen wir R auch noch mitteilen, welche Variable das Zusammenfügen ermöglicht. Diese (in beiden Datensätzen gleiche) Variable heißt urban und wird dem Argument by übergeben. Wir nennen das neue Objekt temp für seine temporäre Nutzung.
temp <- merge(conspiracy, y_mean_k, by = 'urban')
dim(temp) # Dimensionen des temporären Datensatzes
## [1] 2451 10
names(temp) # Spaltennamen des temporären Datensatzes
## [1] "urban" "edu" "gender"
## [4] "age" "GM" "GC"
## [7] "EC" "PW" "CI"
## [10] "EC_mean_k"
head(temp) # ersten 6 Zeilen des temporären Datensatzes
## urban edu gender age GM GC
## 1 rural highschool male 19 4 2.666667
## 2 rural not highschool male 16 5 2.666667
## 3 rural college female 74 2 3.000000
## 4 rural highschool male 38 4 1.666667
## 5 rural highschool male 21 5 4.333333
## 6 rural college male 61 1 1.000000
## EC PW CI EC_mean_k
## 1 1.000000 2.666667 3.666667 2.186667
## 2 1.000000 2.333333 3.333333 2.186667
## 3 2.333333 2.666667 3.333333 2.186667
## 4 3.666667 2.000000 3.333333 2.186667
## 5 4.000000 4.000000 4.333333 2.186667
## 6 1.000000 1.000000 1.000000 2.186667
Anhand der Dimensionen können wir sehen, dass unser neuer Datensatz nun eine Variable mehr hat als conspiracy. Anhand der Variablennamen können wir sehen, dass die zusätzliche Spalte genau die ist, die die Mittelwerte pro Gruppe enthält (EC_mean_k). Mit der head()-Funktion gewinnnen wir weitere Eindrücke, gerne können Sie sich lokal den Datensatz auch mit View(temp) anschauen.
Weitere nötige Informationen für die händische Berechnung der Quadratsummen sind der Gesamtmittelwert und auch die Gruppengröße. Diese können mit uns bekannten Funktion einfach bestimmt werden.
# Gesamtmittelwert ermitteln
y_mean_ges <- mean(conspiracy$EC)
y_mean_ges
## [1] 2.203318
# Gruppengrößen ermitteln
n_k <- table(conspiracy$urban)
n_k
##
## rural suburban urban
## 475 1125 851
Nach diesen Vorbereitungen können wir die Quadratsummen $QS_{inn}$ und $QS_{zw}$ berechnen. $QS_{inn}$ beschreibt die quadratischen Abweichungen aller Gruppenmitglieder von ihrem Gruppenmittelwert. Diese beiden Informationen sind in unserem temporären Datensatz temp in den Variablen temp$EC und temp$EC_mean_k festgehalten.
$$QS_{inn} = \sum_{k = 1}^{K} \sum_{i = 1}^{n_k} (y_{ik}-\overline{y_k})^2$$
QS_inn <- sum((temp$EC - temp$EC_mean_k)^2)
Die Berechnung von $QS_{zw}$ benötigt die einzelnen Gruppengrößen, die wir unter n_k abgelegt haben. Diese werden jeweils mit dem quadrierten Unterschied ihres Gruppenmittelwertes vom Gesamtmittelwert multipliziert. Die einzelnen Gruppenmittelwerte sind in y_mean_k abgelgt. y_mean_k[, 2] wird hier so verwendet, da in y_mean_k in der ersten Spalte die Gruppenzugehörigkeiten stehen und in der 2. Spalte die Mittelwerte selbst. Der Gesamtmittelwert liegt in y_mean_ges.
$$QS_{zw} = \sum_{k = 1}^{K} n_k* (\overline{y_k}-\overline{y})^2$$
QS_zw <- sum(n_k * (y_mean_k[, 2] - y_mean_ges)^2)
Zur inferenzstatistischen Prüfung wird der $F$-Test herangezogen. Für die Berechnung brauchen wir die mittleren Quadratsummen $MQS_{zw} = \frac{QS_{zw}}{K-1}$ und $MQS_{inn} = \frac{QS_{inn}}{N-K}$. Dabei steht $N$ für die Anzahl aller Personen in der Stichprobe und $K$ (weiterhin) für die Anzahl an Gruppen. Folglich können die beiden Werten mit diesem Code per Hand bestimmt werden:
N <- nrow(conspiracy) # Stichprobengröße bestimmen
K <- nlevels(conspiracy$urban) # Gruppenanzahl bestimmen
MQS_inn <- QS_inn / (N - K)
MQS_zw <- QS_zw / (K - 1)
Die Funktionalität von nlevels() basiert auf der Annahme, dass wir kein Level ohne Beobachtung in den Daten haben, aber in diesem Fall wäre es auch schon früher zu Problemen gekommen.
Nun können wir den $F$-Wert bestimmen.
$$F_{emp} = \frac{MQS{zw}}{MQS{inn}}$$ Wir erkennen, dass hier einfach die Variation zwischen den Gruppen (Variation der Mittelwerte) relativ zur (zufälligen) Variation innerhalb der Gruppen betrachtet wird. Ist die Variation zwischen den Gruppen relativ zur zufälligen Variation groß, so kann dies nicht durch Zufall passiert sein: die Mittelwerte müssen sich also bei einem großen $F$-Wert unterscheiden.
F_wert <- MQS_zw/MQS_inn
Das Verhältnis der Quadratsummen ist mit $df_1 = K - 1$ und $df_2 = N - K$ $F$-verteilt. Daher wird der $F_{emp}$ mit dem $F_{krit}$ mit $df_1 = K - 1$ (Zählerfreiheitsgraden) und $df_2 = N - K$ (Nennerfreiheitsgraden) verglichen. In R geht das automatisch mit pf (die Verteilungsfunktion/ kumulative Dichtefunktion der $F$-Verteilung). Diese gibt uns den $p$-Wert wieder. Hierbei muss zunächst der $F_{emp}$ angegeben werden, danach $df_1$ und als letztes $df_2$. lower.tail = FALSE bestimmt, dass wir die Wahrscheinlichkeit (Fläche unter der Kurve) für extremere Werte als unseren beobachteten $F_{emp}$ angezeigt bekommen:
pf(F_wert, nlevels(conspiracy$urban)-1, nrow(conspiracy) - nlevels(conspiracy$urban), lower.tail = FALSE)
## [1] 0.03297692
Grafisch gesehen lassen wir uns also die Fläche für den folgenden Bereich der F-Verteilung anzeigen.
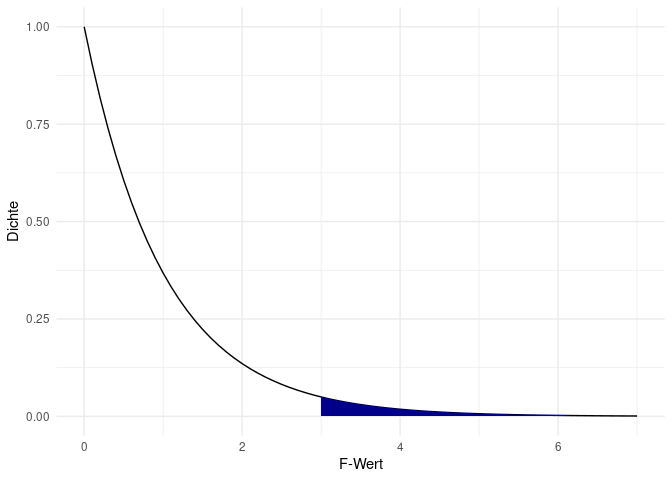
Zur Beurteilung der Signifikanz muss der errechnete p-Wert mit dem vorher festgelegten $\alpha$-Niveau verglichen werden. Da der p-Wert hier niedriger als unser $\alpha$-Niveau von .05 ist, können wir die Nullhypothese verwerfen und die Alternativhypothese annehmen. Bevor wir genauer darauf eingehen, was diese Signifikanzentscheidung bedeutet, schauen wir uns zunächst die Durchführung mithilfe eines Pakets an.
Durchführung per Paket
Da das Ausrechnen per Hand nun doch etwas umständlich ist, bietet R uns einige andere Möglichkeiten. Beispielsweise gibt es anova() oder aov() in der Basisinstallation und diverse weitere Funktionen in Paketen (z. B. Anova() aus car). Bis zum letzten Jahr haben wir die ANOVA hier noch mit dem ez-Paket vorgestellt. Dieses wird jedoch nicht mehr häufig maintained (also Fehler werden nicht behoben) und bietet wenig Expansionsmöglichkeiten zu Kontrasten, besonders im längsschnittlichen Fall. Stattdessen werden wir die ANOVA nun mit dem afex-Paket durchführen. Leider hat auch dieses den Nachteil - Korrekturen der Parameter bei Verletzungen von Voraussetzungen sind nicht komplett implementiert. So ist es also wie so häufig in R - alles hat Vor- und Nachteile. Wir können uns zumindest freuen, dass wir unsere bekannte Schreibweise aus der Regression beibehalten und leicht Kontraste durchführen können. Da wir das Paket bisher nicht genutzt haben, müssen wir es zunächst installieren.
# Paket installieren falls nicht vorhanden
if (!requireNamespace("afex", quietly = TRUE)) {
install.packages("afex")
}
Anschließend kann es geladen werden.
# Paket laden
library(afex)
## Warning: Paket 'afex' wurde unter R Version
## 4.3.2 erstellt
## ************
## Welcome to afex. For support visit: http://afex.singmann.science/
## - Functions for ANOVAs: aov_car(), aov_ez(), and aov_4()
## - Methods for calculating p-values with mixed(): 'S', 'KR', 'LRT', and 'PB'
## - 'afex_aov' and 'mixed' objects can be passed to emmeans() for follow-up tests
## - Get and set global package options with: afex_options()
## - Set sum-to-zero contrasts globally: set_sum_contrasts()
## - For example analyses see: browseVignettes("afex")
## ************
##
## Attache Paket: 'afex'
## Das folgende Objekt ist maskiert 'package:lme4':
##
## lmer
Die Funktion, die wir zur Durchführung der ANOVA nutzen wollen, heißt aov_4(). Wie bereits angekündigt , ist die Syntax ähnlich der der Regressionsanalyse. Die abhängige Variable wird zunächst genannt, dann folgt die Tilde ~, dann die unabhängige Variable und schließlich der Datensatz.
aov_4(EC ~ urban, data = conspiracy)
## Error in if (make.names(name) != name) {: Fehlender Wert, wo TRUE/FALSE nötig ist
Leider funktioniert die Durchführung hier nicht direkt. Die Fehlermeldung spricht an, dass wir einen random-effect in der Formel brauchen. Was das genau ist, besprechen wir in einem späteren Tutorial. Fürs erste können wir das Problem lösen, indem wir eine id-Variable zu unserem Datensatz hinzufügen. Diese macht klar, welche Zeile zu welcher Beobachtung gehört. Bei uns können wir die id einfach durchnummerieren, da jede Beobachtung eine eigene Zeile hat.
conspiracy$id <- 1:nrow(conspiracy)
Die id-Variable muss dann in unserer Regressiongleichung ergänzt werden mit der folgenden Schreibweise, die in einem späteren Tutorial erläutert wird.
aov_4(EC ~ urban + (1|id), data = conspiracy)
## Contrasts set to contr.sum for the following variables: urban
## Anova Table (Type 3 tests)
##
## Response: EC
## Effect df MSE F ges p.value
## 1 urban 2, 2448 1.75 3.42 * .003 .033
## ---
## Signif. codes:
## 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1
Im Output wird zunächst darauf hingewiesen, dass es sich um einen ANOVA Table handelt. Was genau die Klammer - Type 3 tests - bedeutet, besprechen wir in der nächsten Sitzung zur zweifaktoriellen ANOVA. Auch die entstehende Mitteilung besprechen wir dort.
Als zweites wird eine Tabelle angezeigt, die auch dem Output von summary() angewendet auf ein durch lm() erstelltes Objekt ähnlich sieht. Für die uns interessierende Gruppenvariable wird eine Zeile mit allen wichtigen Infos dargestellt. Wir sehen zunächst die Freiheitgrafe (df) für zwischen und innerhalb, die mittlere Quadratsumme der Fehler (MSE), den empirischen $F$-Wert (F) und den $p$-Wert (p.values). Alle diese Werte entsprechen denen, die wir auch händisch bestimmt haben.
Die vorletzte Spalte liefert das generalisierte $\eta^2$ (ges = Generalized Eta-Squared), ein Effektstärkemaß für ANOVAs. Dieses berechnet sich in diesem Fall einfach aus $\eta^2 = \frac{QS_{zw}}{QS_{tot}}$. Für $\eta^2$ haben sich - wie für viele Effektgrößen - Konventionen bezüglich der Interpretation etabliert. Für die Varianzanalyse wird $\eta^2 \approx .01$ als kleiner, $\eta^2 \approx .06$ als mittlerer und $\eta^2 \approx .14$ als großer Effekt interpretiert. Der Wert in unserem Beispiel liegt somit noch unter der Schwelle zu einem kleinen Effekt - die Gruppenunterschiede sind zwar statistisch signifikant von null verschieden, praktisch aber kaum bedeutsam.
Detaillierte Ergebnisse können wir natürlich erhalten, wenn wir die Berechnung in ein Objekt ablegen.
einfakt <- aov_4(EC ~ urban + (1|id), data = conspiracy)
## Contrasts set to contr.sum for the following variables: urban
Es entsteht eine Liste mit 5 Einträgen - klicken Sie die Bestandteile gerne einmal durch (bspw. gibt es noch nähere Informationen zu den Quadratsummen). Wir nutzen das Objekt im Tutorial für den folgenden Abschnitt.
Post-Hoc Analysen
Die ANOVA ist ein Omnibustest - es wird lediglich die Gleichheit aller Gruppen geprüft. Wenn die Nullhypothese verworfen wird, geben die Ergebnisse zunächst keine Auskunft darüber, welche Gruppen sich unterscheiden. Die detaillierte Untersuchung der Gruppenunterschiede wird in der Post-Hoc-Analyse unternommen.
t-Tests
Die naheliegende Untersuchung wäre hier, alle drei Gruppen mithilfe einfacher $t$-Tests zu vergleichen. Da wir hier nicht nur zwei Ausprägungen in unserer Gruppierungsvariable haben, nutzen wir den Befehl pairwise.t.test. Aufgrund der $\alpha$-Fehler Kumulierung müssen die $p$-Werte adjustiert werden (p.adjust = 'bonferroni'). Dabei ist die Bonferroni-Korrektur einer der einfachsten (und gleichzeitig konservativsten) Ansätze: $\alpha_{\text{kor}} = \frac{\alpha}{m}$, wobei $m = \binom{K}{2}$ die Anzahl der durchgeführten Tests ist.
pairwise.t.test(conspiracy$EC, conspiracy$urban, p.adjust = 'bonferroni')
##
## Pairwise comparisons using t tests with pooled SD
##
## data: conspiracy$EC and conspiracy$urban
##
## rural suburban
## suburban 1.000 -
## urban 0.446 0.028
##
## P value adjustment method: bonferroni
Jeder Eintrag in der Tabelle entspricht dann dem korrigierten $p$-Wert von einem Gruppenvergleich. Daher zeigt sich, dass sich ausschließlich Personen aus urban und suburban Umgegbungen in ihrer Überzeugung bezüglich des Extraterrestrial Cover-Ups unterscheiden, da nur hier ein Wert von $p$ < .05($$alpha$) vorliegt.
Tukey Test
Ein Ansatz mit mehr Power (also weniger konservativ) als die einfachen $t$-Tests bietet Tukeys Honest Significant Difference (auch Tukey-Test genannt). Um diesen durchzuführen, brauchen wir ein Objekt des Typens emmGrid bzw. emmeans. Dafür benötigen wir zunächst ein weiteres Paket - das passenderweise auch emmeans heißt. Zunächst installieren wir wieder.
# Paket installieren falls nicht vorhanden
if (!requireNamespace("emmeans", quietly = TRUE)) {
install.packages("emmeans")
}
Nun können wir das Paket einladen.
library(emmeans)
## Warning: Paket 'emmeans' wurde unter R
## Version 4.3.2 erstellt
Nun wandeln wir das Objekt um. Dafür brauchen wir die Funktion emmeans() (die wiederum also genauso heißt wie das Paket). Diese benötigt als erstes Argument unser Objekt und als zweites dann nochmal unsere Gruppierungsvariable mit einer Tilde ~ vorweg. Das erscheint etwas redundant, aber wenn man bspw. mehrere Gruppierungsvariablen hätte, könnte man hier reduzieren.
emm_einfakt <- emmeans(einfakt, ~ urban)
Der Tukey kann dann mit der Funktion pairs() durchgeführt wird. Neben dem Objekt wird als Input noch benötigt, wie wir den p-Wert anpassen adjust wollen. Hier wählen wir tukey. Wir legen das Ergebnis wieder in ein Objekt ab und lassen uns das Ergebnis anzeigen.
tukey <- pairs(emm_einfakt, adjust = "tukey")
tukey
## contrast estimate SE df
## rural - suburban 0.0465 0.0724 2448
## rural - urban -0.1095 0.0757 2448
## suburban - urban -0.1560 0.0601 2448
## t.ratio p.value
## 0.643 0.7964
## -1.445 0.3179
## -2.596 0.0257
##
## P value adjustment: tukey method for comparing a family of 3 estimates
Das zeigt uns für alle 3 Gruppenvergleiche die Mittelwertsdifferenz in estimate an. Die Ergebnisse für die Tukey-korrigierten $p$-Werte sind in der Spalte p.value zu finden. Hier wird deutlich, dass nur der Unterschied zwischen urban und suburban signifikant ist.
Korrigierte Konfidenzintervalle für die Mittelwertsdifferenzen kann man mit confint() anzeigen lassen.
confint(tukey)
## contrast estimate SE df
## rural - suburban 0.0465 0.0724 2448
## rural - urban -0.1095 0.0757 2448
## suburban - urban -0.1560 0.0601 2448
## lower.CL upper.CL
## -0.123 0.2162
## -0.287 0.0682
## -0.297 -0.0151
##
## Confidence level used: 0.95
## Conf-level adjustment: tukey method for comparing a family of 3 estimates
Schließt das Konfidenzintervall für die Mittelwertsdifferenz die 0 ein, so ist diese Mittelwertsdifferenz statistisch nicht signifikant! Als Fazit lässt sich ziehen: In unserer Stichprobe kam es zu Mittelwertsunterschieden auf EC, da sich die Gruppen urban (städtisch) und suburban (vorstädtisch) hinsichtlich der Zustimmung zur Überzeugung, dass die Existenz von Außerirdischen geheimgehalten wird, unterscheiden.
Auch ein hübscher Plot für die Berichterstattung lässt sich erzeugen, der die eben bestimmten Konfidenzintervalle abbildet.
plot(tukey)
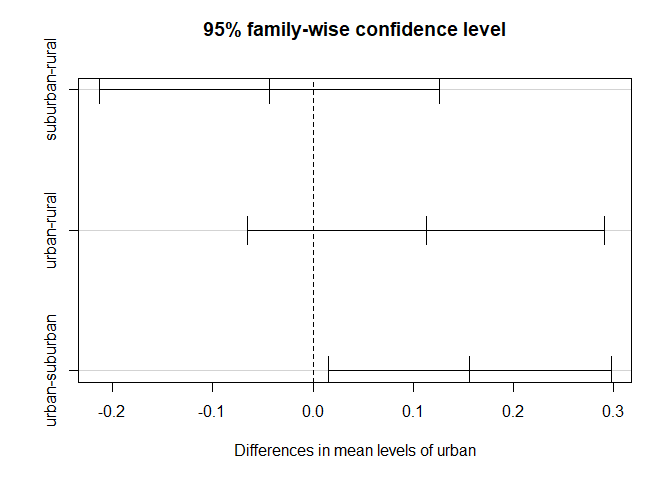
Tipp: Man kann diese Grafik auch noch mit den bereits erlernten ggplot2-Funktionen anpassen.
Fazit
In diesem Tutorial haben wir gelernt, wie die einfaktorielle ANOVA in R durchgeführt werden kann. Neben der händischen Möglichkeit gibt es eine Vielzahl von Paketen, die Möglichkeiten dafür bieten. Wir haben uns dabei für das afex-Paket entschieden, das wir auch in den nächsten Tutorials nutzen werden. In der Funktion sind uns noch einige Bestandteile unklar, aber auch diese werden wir in den nächsten Tutorials klären. Abschließend haben wir auch bereits einen ersten Blick in die Post-Hoc-Analysen geworfen. Hierbei haben wir uns zunächst mit den $t$-Tests beschäftigt, die wir dann mit dem Tukey-Test ergänzt haben. In den nächsten Wochen werden wir noch spezifischere Fragestellungen mit Kontrasten beantworten.
Appendix A
Voraussetzungen der einfaktoriellen ANOVA
Es werden drei Voraussetzungen für die Anwendung einer ANOVA vorgegeben:
- Unabhängigkeit der Residuen
- Homoskedastizität
- Normalverteilung
1) Unabhängigkeit der Residuen
Die Unabhängigkeit der Residuen wäre dann verletzt, wenn abhängige Stichproben vorliegen oder die Stichproben in den Gruppen nicht zufällig zustandegekommen sind (keine Randomisierung). Dies muss also beachtet werden. In unserem Beispiel sind die Stichproben über die Wohnorte hinweg wahrscheinlich nicht abhängig voneinander. Es kann zwar familiäre Bezüge geben, jedoch sollten deren Effekte eher gering ausfallen. Auch die Residuen innerhalb der Gruppen sollten unabhängig sein, da die Personen nicht bewusst einem bestimmten Wohnort zugeordnet wurden, sondern sich dies durch viele Faktoren ergibt. Daher sollte in unserem Beispiel die Unabhängigkeit der Residuen gegeben sein. Wie Sie merken, ist die Unabhängigkeitsannahme schwer zu prüfen und wird daher so gut wie immer durch das Design der Studie “gewährleistet” - bspw. durch Randomisierung.
2) Homoskedastizität
Die Homoskedastizitätsannahme besagt, dass die Varianzen jeder Gruppe über die Gruppen hinweg gleich sind. Deshalb wird diese Annahme auch häufig “Varianzhomogenitätsannahme” genannt. Zur Überprüfung der Homoskedastizität kann der Levene-Test herangezogen werden. Dieser kann mithilfe des car-Pakets angefordert werden. Dazu laden wir zunächst das Paket und führen anschließend die Funktion leveneTest aus. Installiert haben wir das Paket bereits in vorherigen Tutorials und dort auch bereits angewendet.
library(car)
## Lade nötiges Paket: carData
##
## Attache Paket: 'car'
## Das folgende Objekt ist maskiert 'package:dplyr':
##
## recode
## Das folgende Objekt ist maskiert 'package:metafor':
##
## vif
leveneTest(conspiracy$EC ~ conspiracy$urban)
## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 2 2.7631 0.06329 .
## 2448
## ---
## Signif. codes:
## 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Die Funktion nimmt die Variable selbst entgegen sowie die Gruppierungsvariable. EC aus dem conspiracy-Datensatz stellt hierbei die AV dar, die Gruppierungsvariable urban ist die UV. Wir erkennen im Output, was genau der Levene-Test eigentlich macht: Levene's Test for Homogeneity of Variance, nämlich die Varianzen auf Homogenität prüfen. Das Ergebnis ist nicht signifikant. In diesem Fall muss die Annahme der Varianzhomogenität über die drei Gruppen hinweg also nicht verworfen werden.
3) Normalverteilung
Innerhalb jeder Gruppe sollte eine Normalverteilung vorliegen. Diese Annahme bezieht sich, entgegen häufiger Vermutung, auf die Residuen der ANOVA. Wir erkennen diese Annahme aus der Regressionsanalyse wieder, wo wir ebenfalls die Normalverteilung der Residuen, behandelt in Regression III, annahmen, um Inferenzstatistik zu betreiben. Um auf die Residuen einer ANOVA zuzugreifen, kann der Befehl residuals oder resid auf das von uns erstellte Objekt angewendet werden. Wie legen diese mal zusätzlich im Datensatz ab.
conspiracy$resid <- resid(einfakt)
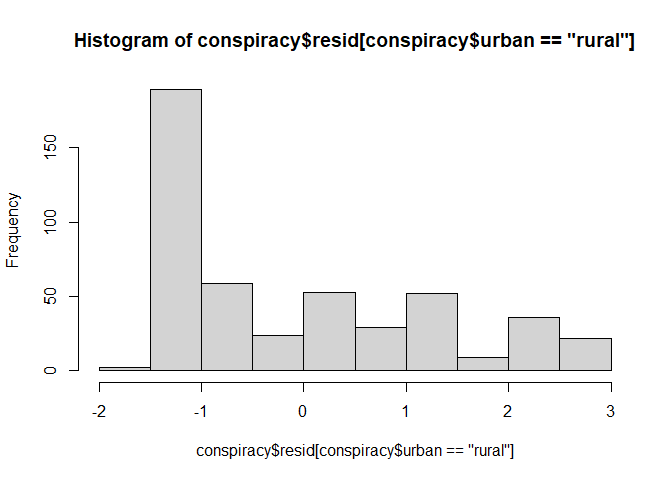
Nun können wir auf die Residuen einzelner Gruppen zugreifen und uns beispielsweise im Histogramm anschauen.
hist(conspiracy$resid[conspiracy$urban == "rural"])
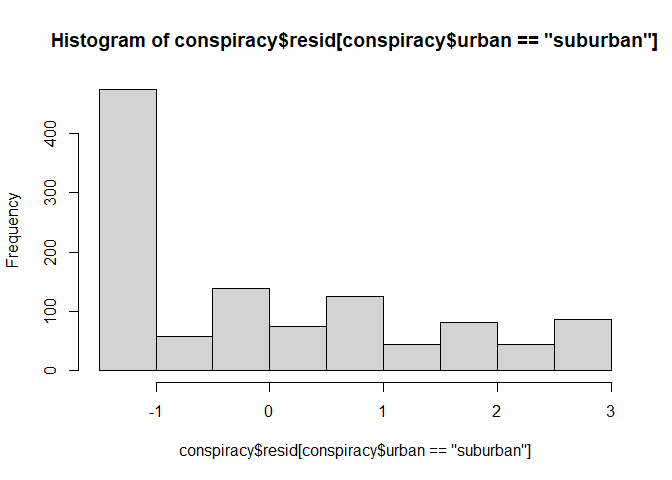
hist(conspiracy$resid[conspiracy$urban == "suburban"])
hist(conspiracy$resid[conspiracy$urban == "urban"])
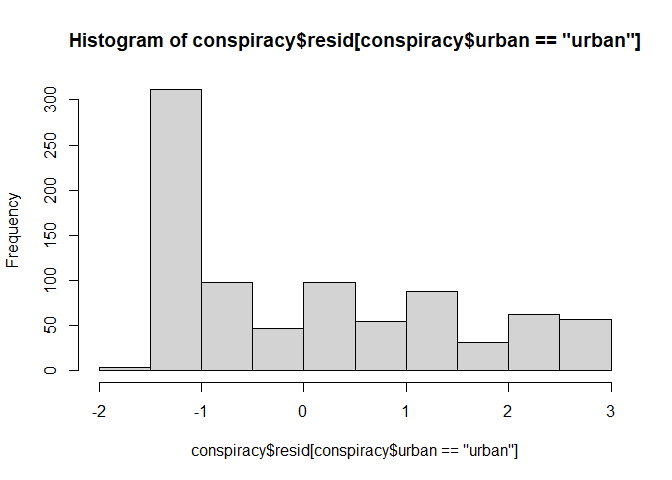
Die Normalverteilung der Residuen scheint schwierig zu sein.
Literatur
Eid, M., Gollwitzer, M., & Schmitt, M. (2017). Statistik und Forschungsmethoden (5. Auflage, 1. Auflage: 2010). Weinheim: Beltz.
- Blau hinterlegte Autor:innenangaben führen Sie direkt zur universitätsinternen Ressource.
](/media/header/earth_and_moon_space.jpg)